Frecuencia del polimorfismo PD-1.3 del gen del receptor de muerte celular programada 1 (PD-1) y su asociación con diabetes tipo 1
Karla Vásquez O.1, Johana Soto S.1, Carolina Pizarro A.1, Francisca Salas P.1 y Francisco Pérez B.1
Frequency of polymorphism PD-1.3 of programmed cell death 1 (PD-1) and their association with type 1 diabetes
1Laboratorio de Genómica Nutricional, Departamento de Nutrición, Facultad de Medicina, Universidad de Chile.
Correspondencia:
Francisco Pérez Bravo
Laboratorio de Genómica
Nutricional,
Departamento de Nutrición,
Facultad de Medicina,
Universidad de Chile.
Avenida Independencia 1027
(Comuna de Independencia),
Santiago, Chile.
E-mail: fperez@med.uchile.cl
Recibido: 27 de noviembre de 2012
Aceptado: 20 de marzo de 2013
Background: The programmed cell death 1 (PDCD-1) immune-receptor is a key element in the negative regulation of peripheral tolerance in T cells. The gene has several polymorphisms and can be associated with susceptibility to autoimmune diseases. Aim: To analyze the frequency and distribution of PD-1.3 polymorphism of PDCD-1 gene and explore its possible contribution as a susceptibility gene for type 1 diabetes (T1D). Patients and Methods: We analyzed 248 cases with T1D with recent diagnosis and 160 control children under 15 years of Santiago. Genetic polymorphism in PD-1 gene variant for PD-1.3 (rs 11568821) was analyzed by polymerase chain reaction and restriction fragment length polymorphism. Comparison of genotype, allele frequency and consistency with respect to Hardy-Weinberg were analyzed using χ2 tests and Fisher exact test. Results: There was a very low frequency of the genotype A/A, both in T1D patients and in controls (< 2%). The A/G genotype was more common in diabetic patients than in controls (41.6 and 18.8% respectively, p < 0.04). G/G genotype was more common in controls than in patients (79.4 and 56.8% respectively, p < 0.02). T1D patients carrying genotype G/G had a higher frequency of anti-GAD65 and anti-A-2 antibodies (81 and 67% respectively). Conclusions: The distribution of PD-1.3 genotype frequencies are similar to that reported elsewhere. Possibly, this genetic variant (rs 11568821) does not have an important marker role in Chilean T1D patients.
Key words: Type 1 diabetes, PDCD1 gene polymorphisms, genetic markers, autoimmunity.
La diabetes mellitus tipo 1 (DM1) constituye el trastorno endocrino-metabólico más común en la infancia y adolescencia. Su etiología es compleja e intervienen factores ambientales que interactúan con componentes de predisposición genética, lo que finalmente gatilla un irreversible proceso autoinmune que destruye las células β pancreáticas1. Entre los múltiples factores ambientales que se han relacionado a la DM1 se encuentran las infecciones virales, algunos agentes dietarios y la hipótesis de la higienes2-7. Todos ellos conducen de alguna forma, aún desconocida, a la ruptura de la tolerancia autogénica normal generando autoinmunidad contra la célula β pancreática8. Entre los factores genéticos asociados a la DM1, el sistema mayor de histocompatibilidad (HLA de clase II) ha sido el candidato principal desde hace más de tres décadas debido al papel central que juega en el proceso de reconocimiento antigénico9. Además de esta región HLA, se han identificado y propuesto otras 21 regiones candidatas como posibles marcadores de susceptibilidad a la DM110. Parte de los estudios de los últimos años, han focalizado su atención en aquellos genes de regulación negativa de la respuesta inmune, es decir aquellos que operan frenando la respuesta, una vez que el antígeno ha sido destruido. Se han identificado tres regiones importantes en esta regulación negativa de los linfocitos T: CTLA-4, PTPN22 y PD-1. Para CTLA-4 existen importantes estudios que han revelado su papel como variante de susceptibilidad en diversas patologías autoinmunes11,12, el gen PTPN22 que codifica para la Proteína Tirosina-Fosfatasa (LYP) también ha demostrado relevancia a través de la regulación de la respuesta del linfocito T13 y finalmente el gen que codifica para los receptores de muerte celular programada 1 (PD-1) el cual participa también en el reconocimiento de estructuras que regulan negativamente la relación entre la célula presentadora del antígeno (célula β pancreática en el caso de la DM1) y el linfocito T14,15.
El gen PD-1 y sus proteínas ligando tienen un importante rol en la inducción y/o mantención de la tolerancia periférica. A través de la interacción con sus ligandos (PDL1 y PDL2), se inhibe la estimulación antigénica de las células T y B, estos ligandos pertenecen a la familia de proteínas B7 y se expresan en órganos linfoides, como no linfoides. El balance de señales inhibitorias (PD-1 y CTLA-4) y de estimulación (B7) genera interacciones entre la superficie celular de los linfocitos T y las células presentadoras de antígenos y corresponden a mecanismos cruciales de la homeostasis celular de la tolerancia periférica16. El gen humano para PD-1, localizado en el cromosoma 2q37 presenta varios polimorfismos de nucleótido simple (SNPs) asociados con autoinmunidad: Lupus eritematoso sistémico (LES), artritis reumatoidea (AR), tiroiditis y DM117. Los estudios de polimorfismo genético en pacientes con DM1 han mostrado resultados controversiales según la población analizada18-21. El objetivo de esta investigación fue analizar la frecuencia y posible asociación del polimorfismo PD-1.3 con la DM1 en un grupo de pacientes chilenos.
Pacientes y Métodos
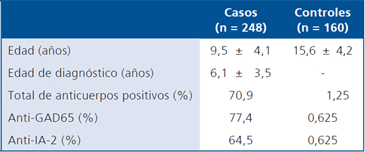
El estudio incluyó 248 pacientes con DM1 (niños y adolescentes)
y 160 controles adolescentes aparentemente sanos,
sin antecedentes familiares de diabetes u otras enfermedades
autoinmunes (datos obtenidos mediante encuesta familiar).
Las características clínicas de ambos grupos se resumen en
la Tabla 1. Los controles fueron contactados de dos colegios
de Santiago (comunas de Independencia y Macul), por lo que
estimamos que son comparables en términos de su estatus socio-
demográfico respecto de los casos con DM1 que provienen
del IDIMI, Hospital San Borja Arriarán. El protocolo de
esta investigación ha sido aprobado por los comités de ética
respectivos (Facultad de Medicina, Universidad de Chile y Servicio de Salud Metropolitano Central, Hospital San Borja Arriarán). Todos los pacientes, controles y/o sus padres o
tutores entregaron su consentimiento por escrito.
Extracción de ADN y determinación de polimorfismos PD-1
El ADN genómico se extrajo a partir de una muestra de sangre periférica (3 ml) utilizando técnicas estándares de lisis celular (Winkler, Santiago, Chile). El polimorfismo PD- 1.3 (rs11568821) se determinó a través de partidores específicos (Forward: 5´TGA TCC ACT GTG CCT TCC TTC CTG 3´; Reverse: 5´CCT CTG AAA TGT CCC TGG CAT TCT 3´) y cada reacción de PCR fue realizada en 25 μL de volumen de reacción, conteniendo 10 μM de cada partidor, 11 μL de ADN de cada una de las muestras, 10 mM de dNTP, 5 μL de Buffer NH4SO4, 3 μL de Mg+2 25 mM, 0,2 μL de Taq- Polimerasa y H2O. La reacción se estandarizó en 35 ciclos a fin de desnaturar el ADN (90ºC), unir los partidores (58ºC) y realizar la extensión (72ºC). El polimorfismo PD-1.3 se determinó a través de fragmentos de restricción (RFLPs) y para ello se tomaron 15 μL de cada producto de PCR que fueron incubados a 37°C durante 1 h con 0,1 U de la enzima Pst1; 5 μL de Buffer NE 10X, 0,5 μL de BSA y 29,5 μL de H20, el producto de digestión fue visualizado en geles de agarosa al 3%.
Análisis serológico de anticuerpos
Los anticuerpos anti-GAD65 y anti-IA2 fueron determinados mediante inmunoensayo enzimático (ELISA) con el uso de kits comerciales (Medizym® Diagnostic, Berlín, Germany). Esta detección de anticuerpos es semi-cuantitativa y establece puntos de corte frente a una curva de calibración considerando, como referencia el valor de 5 UI/mL para GAD65 y 10 UI/mL para IA2. Aquellas muestras con un título mayor o igual a dicho valor fueron consideradas muestras positivas. Se obtuvo una sensibilidad y especificidad del 92,3% y 98,6% para anti-GAD65 y de 75% y 98% para anti-IA-2.
Análisis estadístico
Las comparaciones de frecuencia alélicas y genotípicas entre casos y controles fueron analizadas mediante las pruebas de χ2 y Prueba exacta de Fisher utilizando el programa SHESIS (URL: http://202.120.7.14/analysis/myAnalysis.php). Se evaluó la concordancia de las frecuencias genotípicas con respecto al equilibrio de Hardy-Weinberg por χ2. Las comparaciones de las variables continuas del estudio se realizaron mediante prueba t de Student, la comparación de anticuerpos a través de test de proporciones. Un valor de p < 0,05 fue considerado como estadísticamente significativo.
Resultados
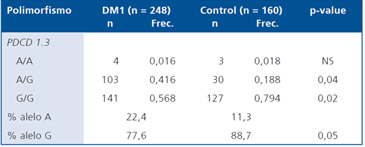
La Tabla 2 muestra la distribución observada para el
polimorfismo PD-1.3. Existe una muy baja frecuencia del
genotipo A/A tanto en pacientes con DM1 como en controles
(1,6% y 1,8% respectivamente). Esta baja frecuencia en concordante con los datos provenientes de otros estudios
donde se informa de la misma baja frecuencia. El genotipo
heterocigoto A/G muestra diferencias al comparar ambos
grupos, 41,6% de los pacientes con DM1 son portadores de
este genotipo vs el 18,8% presente en controles (p < 0,04).
El polimorfismo G/G también mostró diferencias, siendo
muy frecuente en la población control con un 79,4% vs un
56,8% en pacientes con DM1 (p < 0,02). Como consecuencia
de esta distribución genotípica, la frecuencia del alelo G es
significativamente mayor en controles (88,7% vs 77,6%, p <
0,05). Lamentablemente, el análisis de este polimorfismo no
se muestra en equilibrio de Hardy-Weinberg, situación que
también ya ha sido descrita en los estudios publicados para
esta variante.
Finalmente, la Tabla 3 muestra los resultados del estudio de asociación entre genotipos PD-1.3 y marcadores inmunológicos característicos de la DM1. Dada la baja frecuencia del genotipo A/A el resultado es solamente referencial, puesto que su baja distribución impide realizar comparaciones. Sin embargo, para los genotipos A/G y G/G se observa una mayor frecuencia de anticuerpos positivos. El genotipo G/G se asocia a un porcentaje mayor de anti-GAD65 (85,1%) y al mayor porcentaje de anti-IA-2 (66,6%).
Discusión
Dado el importante papel que tienen las moléculas de
inmuno-regulación negativa en la proliferación de células T,
tanto la molécula PD-1 como CTLA-4 han sido objeto de innumerables
estudios en los últimos 10 años con el fin de establecer
su relación con patologías de origen autoinmune11,13,22.
Existe en la literatura un amplio número de publicaciones en
relación a CTLA-4, sin embargo, los estudios que involucran
a PD-1 son de menor cuantía. Al respecto, la información publicada
respecto a la posible asociación de este polimorfismo
en la DM1 es limitada y controversial18-23.
Nuestro estudio mostró que el polimorfismo PD-1.3 y en especial el genotipo G/G no estaría asociado a la enfermedad, ya que éste resultó ser más frecuente entre los controles. No obstante, cuando se analizó este genotipo en el contexto de la autoinmunidad entre los pacientes con DM1, fue posible observar que este genotipo se encuentra asociado a un mayor patrón autoinmune. Esta situación ya ha sido descrita en otras enfermedades autoinmunes24.
Nuestros resultados de distribución del polimorfismo PD-1.3 están más próximos a los observados en población europea, no así a las frecuencias que muestran grupos asiáticos. La muy baja frecuencia observada para uno de los polimorfismos (A/A) también ha sido descrita en diferentes etnias y en distintas patologías autoinmunes tales como la Enfermedad de Graves, Enfermedad de Addison y Lupus25-27.
Hay dos aspectos importantes a considerar en este estudio, uno de ellos es el tamaño de la muestra y su procedencia. Sin embargo, esto ha sido reportado en la mayoría de los estudios.Al respecto, creemos que nuestra muestra no presenta un sesgo de selección, es decir que los pacientes y controles no sean representativos de la población chilena, dado que están seleccionadas de lugares comparables y representan a población pediátrica y adolescente del sector medio-bajo de nuestra población. Sin embargo, la baja frecuencia de una de las variantes genotípicas (A/A) hace predecible un desvío del equilibrio28.
H-W equilibrium p < 0,01.
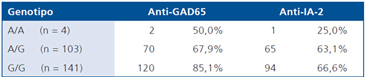
Los resultados obtenidos por este estudio, no mostraron asociación genotípica del polimorfismo PD-1.3 con DM1. No obstante, dentro del grupo con DM1 el genotipo G/G parece relacionarse con una mayor frecuencia de autoinmunidad (mayor frecuencia de anti-GAD y anti-IA-2). El polimorfismo PD-1.3 del gen PDCD1 es uno de los pocos polimorfismos funcionales descritos para este gen, dentro de más de 130 polimorfismos, es decir que la sustitución nucleotídica es capaz de producir un cambio en el marco de lectura de la proteína, esto podría relacionarse tal vez con una mayor reactividad celular (situación no abordada por este estudio) y repercutir en un mayor patrón de autoinmunidad. En este nuevo escenario, los estudios dirigidos a la caracterización de otros polimorfismos funcionales en la regulación negativa de la célula T podrían tener en el futuro una relevancia importante para entender los mecanismos que alteran la tolerancia inmunológica.
Agradecimientos
Los autores agradecen la colaboración de todos los niños
y sus familias participantes en este estudio en especial al
Hospital San Borja-Arriarán. Esta investigación fue financiadacon el proyecto FONDECYT 1100075.
Referencias bibliográficas
- Van Belle TL, Coppieters KT, von Herrath MG. 2011. Type 1 diabetes: etiology, immunology and therapeutic strategies. Physiol Rev 91: 79-118.
- Van der Werf N, Kroese F, Rozing J, Hillebrands JL. 2007. Viral infections as potential triggers of type 1 diabetes. Diab Metab Res Rev 23: 169-183.
- Lefebvre D, Powell K, Strom A, Scott F. 2006. Dietary Proteins as Environmental Modifiers of Type 1 Diabetes Mellitus. Ann Rev Nutr 26: 175-202.
- Gale EA. 2002. A missing link in the hygiene hypothesis? Diabetologia 45: 588-594.
- Norris JM, Barriga K, Klingensmith G, Hoffman M, Eisenbarth GS, Erlich HA, Rewers M. 2003. Timing of initial cereal exposure in infancy and risk of islet autoimmunity. JAMA 290: 1713-1720.
- MacFarlane AJ, Strom A, Scott FW. 2009. Epigenetics: deciphering how environmental factors may modify autoimmune type 1 diabetes. Mamm Genome 20: 624-632.
- Javierre BM, Hernando H, Ballestar E. 2011. Environmental triggers and epigenetic deregulation in autoimmune disease. Discov Med 12: 535-545.
- Zhang L, Eisenbarth GS. 2011. Prediction and prevention of Type 1 diabetes mellitus. J Diabetes 3: 48-57.
- Pociot F, Akolkar B, Concannon P, Erlich HA, Julier C, Morahan G, et al. 2010. Genetics of Type 1 Diabetes: What’s Next? Diabetes 59: 1561-1571.
- Concannon P, Rich S, Nepom GT. 2009. Genetics of type 1A diabetes. N Engl J Med 360: 1646-1651.
- Vaidya B, Pearce S. 2004. The emerging role of the CTLA-4 gene in autoimmne endocrinophaties. Eur J Endocrinol 150: 619-626.
- Ueda H, Howson JM, Espósito L, Heward J, Snook H, Chamberlain G, et al. 2003. Association of the T-cell regulatory gene CTLA-4 with susceptibility to autoimmune disease. Nature 423: 506-511.
- Vang T, Miletic AV, Arimura Y, Tautz L, Rickert RC, Mustelin T. 2008. Protein tyrosine phosphatases in autoimmunity. Ann Rev Autoimmunity 26: 29-55.
- Keir ME, Butte MJ, Freeman GJ, Sharpe AH. 2008. PD-1 and its ligands in tolerance and immunity. Ann Rev Immunol 26: 677-704.
- Sharpe AH, Wherry EJ, Ahmed R, Freeman GJ. 2007. The function of programmed cell death 1 and its ligands in regulatin autoimmunity and infection. Nat Immunol 8: 239-245.
- Okazaki T, Honjo T. 2007. PD-1 and PD-1 ligands: from discovery to clinical application. Int Immunol 19: 813-824.
- Okazaki T, Honjo T. 2006. The PD-1-PD-L pathway in immunological tolerance. TRENDS in immunology 27: 195-201.
- Asad S, Nikamo P, Torn C, Landin-Olsson M, Lernmark A, Alarcón-Riquelme M, Kockum I. 2007. No evidence of association of the PDCD1 gene with type 1 diabetes. Diabet Med 24: 1473-1477.
- Nielsen C, Hansen D, Husby S, Jacobsen BB, Lillevang ST. 2003. Association of a putative regulatory polymorphism in the PD-1 gene with susceptibility to type 1 diabetes. Tissue Antigens 62: 492-497.
- Ni R, Ihara K, Miyako K, Kuromaru R, Inuo M, Kohno H, Hara T. 2007. PD-1 gene haplotype is associated with the development of type 1 diabetes mellitus in Japanese children. Hum Genet 121: 223-232.
- Hiromine Y, Ikegami H, Fujisawa T, Nojima K, Kawabata Y, Noso S, et al. 2007. Trinucleotide repeats of programmed cell death-1 gene is associated with susceptibility to type 1 diabetes mellitus. Metabolism 56: 905-909.
- Riley J, June C. 2006. The road to recovery: translating PD-1 biology into clinical benefit. TRENDS in immunology 28: 48-50.
- Fichna M, Zurawek M, Januszkiewicz-Lewandowska D, Fichna P, Nowak J. 2010. PTPN22, PDCD1 and CYP27B1 polymorphisms and susceptibility to type 1 diabetes in Polish patients. Int J Immunogenetics 37: 367-372.
- Juran BD, Atkinson EJ, Schlicht EM, Fridley BL, Petersen GM, Lazaridis KN. 2008. Interacting alleles of the coinhibitory immunoreceptor genes cytotoxic T-lymphocyte antigen 4 and programmed cell-death 1 influence risk and features of primary biliary cirrhosis. Hepatology 47: 563-574.
- Lin SC, Yen JH, Tsai JJ, Tsai WC, Ou TT, Liu HW, Chen CJ. 2004. Association of a programmed death 1 gene polymorphism with the development of rheumatoid arthritis, but not systemic lupus erythematosus. Arthritis & Rheumatism 50: 770-775.
- Thorburn CM, Prokunina-Olsson L, Sterba KA, Lum RF, Seldin MF, Alarcón-Riquelme ME, Criswell LA. 2007. Association of PDCD1 genetic variation with risk and clinical manifestations of systemic lupus erythematosus in a multiethnic cohort. Genes Immunol 8: 279-287.
- Sutherland A, Davies J, Owen CJ, Vaikkakara S, Walker T, Cheetham D, et al. 2010. Genomic polymorphism at the interferon induced helicase (IF1H1) locus contributes to Grave´s disease susceptibility. J Clin Endocrinol Metab 92: 3338-3341.
- Flores S, Beems M, Oyarzún A, Carrasco E, Pérez-Bravo F. 2010. Programmed cell death 1 (PDCD1) gene polymorphisms and type 1 diabetes in Chilean children. Rev Med Chile 138: 543-550.