Nuevas perspectivas en la genética de la Diabetes Mellitus tipo 2 y sus complicaciones crónicas
Daniela Seelenfreund H.1a, Sergio Lobos C.1a y Pilar Durruty A.2a
New perspectives in the genetics of diabetes mellitus and its complications
1Departamento de Bioquímica y Biología Molecular, Facultad de Ciencias Químicas y Farmacéuticas, Universidad de Chile.
2 Unidad de Diabetes y Nutrición, Facultad de Medicina Occidente, Universidad de Chile
a Bioquímico
Correspondencia a: Daniela Seelenfreund, Departamento de Bioquímica y Biología Molecular, Facultad de Ciencias Químicas y Farmacéuticas, Universidad de Chile. Vicuña Mackenna 20, Providencia, Santiago, Chile. e-mail: dseelen@ciq.uchile.cl
This review describes the advances in the knowledge about the genetic aspects of common chronic complications of diabetes with prognostic significance, such as diabetic nephropathy and cardiovascular diseases. It is well known that the genetic factors responsible for chronic complications are different from those that cause Diabetes Mellitus. Until recently, the studies were limited to the analysis of individual genes associated or related to multifactorial diseases. However at the present time the “genome wide association studies” lead to a great advance in knowledge. The analysis of genetic variations or polymorphisms allows the understanding of human individuality and the predisposition towards certain diseases. A new research field appeared in 2004, when small messenger RNAs, called microRNA related to Diabetes Mellitus and its chronic complications, were identified. The function of these RNAs is to regulate several target genes. These affect insulin secretion and action and genes related to microangiopathic and specific macroangiopahic complications. This new knowledge will identify new genes related to the disease and will allow the development of therapeutic strategies devised according to individual susceptibility towards specific chronic complication.
La actual pandemia de diabetes mellitus (DM) obliga a dirigir los esfuerzos hacia la prevención y disminución de la progresión de la enfermedad y sus complicaciones. Una forma relevante de abordar estos problemas es la identificación de los genes involucrados en la presentación y desarrollo de la DM y de las complicaciones crónicas, pues este conocimiento permitirá la implementación de estrategias terapéuticas específicas.
El avance de las nuevas herramientas genéticas, tales como el análisis de las variaciones de genomas completos (“Genome wide association” o estudios GWA) ameritan una revisión de los conocimientos alcanzados. Además, durante la última década se ha descubierto otra clase de moléculas que regulan la expresión de los genes, un tipo de ácidos ribonucleicos pequeños (RNAs) denominados microRNAs que presentan un gran potencial en el estudio genético de distintas patologías. Con el objeto de ilustrar a los lectores de su eventual importancia en la investigación de una enfermedad tan frecuente como la DM, describimos los avances recientes en este campo en relación a esta patología y a las complicaciones de mayor morbi-mortalidad, como son la Nefropatía Diabética (ND) y la Enfermedad Cardiovascular (ECV).
Impacto de la DM y sus complicaciones crónicas
Actualmente, el Ministerio de Salud de Chile estima que la frecuencia de DM en el país es de 4,3%, cifra inferior al 5,3% encontrada en 19781. Otros estudios recientes han informado porcentajes cercanos al 5,39%2. Cualquiera sea la prevalencia real de la DM, ésta irá en aumento por el envejecimiento de nuestra población y la tendencia creciente al sobrepeso. Desde sus etapas iniciales, como es la intolerancia a la glucosa o prediabetes, el paciente puede desarrollar complicaciones crónicas macrovasculares y posteriormente microvasculares, patologías que disminuyen la expectativa de vida de estos enfermos.
Complicaciones crónicas de diabetes mellitus tipo 1 (DM1) y tipo 2 (DM2)
Las complicaciones de la DM1 y DM2 son básicamente las mismas. En ambas patologías las más frecuentes son las microangiopáticas: ND, retinopatía y neuropatía diabética, y macroangiopáticas, que son la ECV, vascular encefálica y vascular periférica.
La ND es la complicación microangiopática más frecuente de la DM, presentándose en el 20-40% de los pacientes luego de 10 años de evolución natural. A pesar de los tratamientos actuales, el 20% de los DM2 y el 50% de los DM1 con ND desarrollan insuficiencia renal (IR)3. Por otra parte, los diabéticos nefrópatas presentan una frecuencia 8 a 15 veces mayor de ECV y mortalidad cardiovascular 37 veces superior a la de la población general4.
En ambos tipos de diabetes la prevalencia y gravedad de la ECV es significativamente mayor que en los no diabéticos. En los últimos 40 años la mortalidad coronaria ha disminuido en la población general, pero no en los diabéticos5. La enfermedad coronaria es la principal causa de muerte tanto en DM1 como en DM2, siendo la hiperglicemia directamente un factor de riesgo de mortalidad.
La frecuencia de enfermedad coronaria en los diabéticos está directamente relacionada con la prevalencia de la enfermedad en la población general, alta en algunas etnias y baja en otras (Japón y China), lo que pone de manifiesto su base genética. La influencia ambiental se manifiesta cuando en individuos de bajo riesgo aumenta la frecuencia de la enfermedad al ser trasladados a lugares de alta prevalencia.
El factor genético en la etiopatogenia de la DM tipo 2
En el desarrollo de la DM2 es necesaria la participación de factores genéticos y ambientales, tales como la obesidad, la obesidad androide y el sedentarismo que determinan alteraciones en la secreción de insulina al estímulo con glucosa y en la acción de la hormona en los tejidos periféricos.
En estudios de agregación familiar se ha encontrado un 10-30% de DM en los familiares de los pacientes diabéticos, y cifras de 1-6% en los parientes de los sujetos sin esta patología6. La historia familiar es un factor de riesgo independiente para las enfermedades crónicas, incluyendo la DM2. En EE.UU. el 88-95% de los adultos diabéticos tiene algún pariente de primer grado con DM2, cifra que baja al 70-77% en los familiares de segundo grado7. Se ha encontrado que el tener diabetes en la familia permite identificar personas con diabetes no diagnosticada, con mayor sensibilidad que la obesidad8.
En gemelos monocigóticos hay una concordancia de DM2 de 70% y de 37% en mellizos dicigóticos9. En los países desarrollados, con factores ambientales que favorecen la DM, la concordancia en los gemelos puede llegar al 100%, lo que pone de manifiesto la importancia primaria de la susceptibilidad genética a la enfermedad.
La DM2 presenta una gran variabilidad étnica, en 1998 en EE.UU., la enfermedad se presentaba con una prevalencia de 10,8% en los blancos no hispánicos y 64,6% en los indios Pima de Arizona10. Las poblaciones asiáticas y los grupos indígenas de América tienen una baja prevalencia de DM2. En los últimos 20 años, en nuestro país, la prevalencia de DM2 ha aumentado en aimaras y en mapuches, alcanzando cifras de 1,5% y 3,7% respectivamente, manteniéndose dentro de las cifras más bajas de los registros internacionales11,12.
Aunque en la etiopatogenia de la DM2 el factor genético es importante, no se conocen aún los genes que están involucrados y los hallazgos hasta ahora apuntan a que su genética es muy compleja.
La DM2 parece deberse a múltiples genes, de los cuales cada uno por sí mismo sólo tiene un impacto menor13. Se han postulado diversos genes candidatos, los cuales han sido difíciles de confirmar, en parte por el sesgo en la selección y tamaño de la población analizada y las variaciones étnicas. Además, es probable que lo que en conjunto se denomina o que arbitrariamente se define como DM2, corresponda a diversas patologías sobrepuestas con una sintomatología común.
Las estrategias tradicionales para el estudio genético se basan en establecer una asociación entre el gen candidato y un fenotipo dado, de un seguimiento de familias, mellizos y hermanos o de casos/controles pareados afectados por la patología. Estos análisis han permitido establecer que la DM1 tiene una asociación familiar y alteraciones en los genes que codifican para determinados antígenos HLA. En DM2 los resultados han sido mucho más controvertidos. Más aún, los estudios en general analizan sólo regiones codificantes, obviando la participación de secuencias regulatorias, tanto de regiones promotoras como de secuencias involucradas en el procesamiento del pre-mRNA eucariótico (“splicing”), las cuales también podrían jugar un papel importante.
El factor genético en las complicaciones crónicas de la DM
Los factores que participan en la etiología de la DM2 son diferentes a los que conducen a las complicaciones crónicas. No todos los pacientes diabéticos presentan complicaciones, ni con la misma severidad, por lo que se ha sugerido que existirían variantes genéticas que explicarían estas diferencias. La ND es altamente heredable en algunas familias y ciertas etnias14, sugiriendo que los genes de susceptibilidad de daño renal son diferentes a los factores genéticos que causan la DM215. Los estudios prospectivos sugieren que la hiperglicemia y la hipertensión son factores importantes, pero no suficientes para el desarrollo de las complicaciones crónicas, siendo necesaria la susceptibilidad genética. También estaría determinada genéticamente la progresión de ND a IR, dado que no todos los nefrópatas evolucionan a IR y, además, la respuesta al tratamiento con ARA II o con IECA es variable en los distintos individuos; en algunos casos estos fármacos son capaces de retardar o incluso detener la progresión de la enfermedad, pero en ninguno de ellos se logra su norma lización16.
Es probable que el riesgo de nefropatía diabética se deba a los alelos heredados de riesgo en los loci de susceptibilidad que se ponen de manifiesto frente a la hiperglicemia, cuyo análisis en el futuro permitirá diseñar nuevas terapias que retarden o detengan el desarrollo de IR.
Es evidente que si se puede identificar a las personas diabéticas con probabilidad de desarrollar daño renal y/o coronario y tomar medidas para prevenirlas, se obtendrá una mejor calidad de vida de los pacientes y una disminución considerable de los costos en salud (Figura 1). A su vez, será posible evitar tratamientos innecesarios en los individuos que tienen diabetes, pero con poco o ningún riesgo de ND. Esta es una perspectiva futura promisoria, y la ND podría ser pionera en la aplicación de la medicina personalizada17.
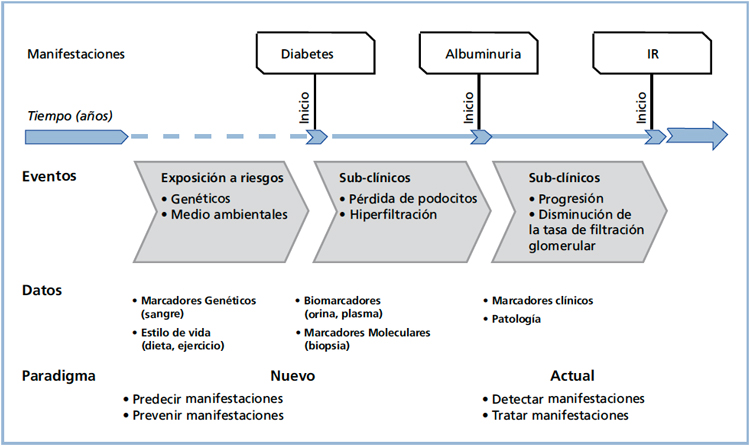
Figura 1. Medicina personalizada en la Nefropatía Diabética.
Adaptado de Susztak y Böttinger17
Investigación genética en DM
Investigación genética en DM Las nuevas tecnologías son una extensión del análisis de asociación a nivel de genomas completos o GWA. Éstas presentan la ventaja de que se realizan sin sesgo, pues no parten de la premisa de que el gen en estudio tiene (o no) una relación con la patología, sino que corresponden al tamizado de miles de variantes genéticas de la secuencia completa del genoma humano. Este tipo de estudios se basa en el desarrollo de plataformas de secuenciación automatizada, la disponibilidad de bases de datos de polimorfismos de un solo nucleótido o SNPs y haplotipos (combinaciones de SNPs), las nuevas herramientas de análisis estadístico y la posibilidad de formar grandes cohortes con miles de individuos13. Los SNPs son las variaciones genéticas más frecuentes en el genoma humano, pues se conocen ya más de 1.000.000 de SNPs diferentes, los que contribuyen en forma importante a las diferencias fenotípicas entre individuos18.
Los barridos genómicos GWA comparan genotipos entre casos y controles con el objeto de identificar genes de susceptibilidad que presenten mayor frecuencia en los pacientes. A la fecha se han realizado varias investigaciones de tipo GWA en DM1 y DM2, lo cual permite la integración y comparación de datos19-21. Los análisis de GWA en DM1 han confirmado que los genes más importantes en su etiología son el gen que codifica para el locus IDDM2, así como el gen IDDM1 del complejo HLA19. Nuevas regiones ligadas a DM1, de acuerdo a estudios GWA, son genes de la región 2q31-q33 como IDDM7 e IDDM12, así como otras 6 regiones cromosómicas19.
En DM2 las investigaciones clásicas de asociación han identificado genes relacionados con la secreción de insulina (IRS-2 y CAPN10) y la resistencia a insulina (IRS-1 y PPAR). Los estudios GWA han confirmado la importancia de los genes ya descritos, pero ninguno conlleva un factor de riesgo alto y tampoco se repiten en todas las poblaciones estudiadas. Estos resultados nuevamente sugieren que aún quedan genes de susceptibilidad a DM2 por descubrir20,21.
Un análisis de más de 116.000 SNPs en mexicanos americanos encontró asociación con variantes en genes ya conocidos, con variantes en intrones o regiones no codificantes y también genes que no se habían relacionado con DM2, lo cual confirma la capacidad de los análisis GWA de aportar nuevos loci para el estudio de esta enfermedad (13 SNPs asociados a DM2)20.
El estudio del Wellcome Trust Case Control Consortium (WTCCC) de 50 laboratorios británicos en relación al riesgo genético de 7 enfermedades crónicas, entre ellas la DM1 y la DM2, analizó 500.000 marcadores genéticos en 14.000 pacientes y 3.000 controles21. En DM2 se confirmó la contribución de variantes de los genes PPARG, KCNJ11 (“the inwardly-rectifying Kir6.2 component of the pancreatic beta-cell KATP channel”), y TCF7L2 (“transcription factor7-like 2”). Se descubrieron 2 loci nuevos correspondientes al gen FTO (“Fat-mass and obesity- associated”) y a SNPs del intron 5 del gen CDKAL1 (“CDK5 regulatory subunit-associated protein1-like1”) de función desconocida. También se encontraron 3 loci nuevos asociados a DM121.
Investigación genética en ND y ECV
El análisis genético tradicional no ha identificado genes relacionados con ND. Se ha descrito que la nefrina, una proteína fundamental del diafragma de filtración, se encuentra disminuida en la ND22,23. Esta proteína fue descubierta en 1999 porque sus mutaciones producen el síndrome nefrótico congénito finlandés24. Se han descrito más de 100 SNPs en el gen que codifica para esta proteína, y no está claro si alguna de estas variantes genéticas se relaciona con la ND. Daimon y cols.25 describieron que un SNP en el promotor del gen de la nefrina tiene relación con la intolerancia a la glucosa, aunque no con ND.
¿Cómo saber si alguno de estos SNPs tiene importancia en esta enfermedad? Una estrategia consiste en realizar un análisis bioinformático de los SNPs descritos en el gen de interés descartando los de bajo o nulo riesgo e identificando SNPs que potencialmente podrían ser de riesgo, pues producirían una proteína alterada que sería fundamental en la etiología de la patología. Sin embargo, es importante verificar hasta donde sea posible si el SNP sugerido como de riesgo, genera un producto alterado o causa un efecto in vivo. Por ejemplo, el análisis informático del SNP intrónico rs#466452 del gen de la nefrina sugería un cambio en el procesamiento del transcrito, dado que afectaría un putativo sitio de regulación26. Sin embargo, su análisis en 230 individuos chilenos indicó que este SNP se encuentra con igual frecuencia en pacientes diabéticos y controles sanos, y no tendría relación con el desarrollo ni la progresión de ND en pacientes diabéticos tipo 1 o tipo 227. Más aún, el análisis directo del mRNA de nefrina aislado de una biopsia renal obtenida de un paciente que presentaba la variante genética del SNP rs#466452, indicó que el procesamiento de dicho transcrito era normal27.
Los pacientes con ND presentan mayor riesgo de ECV, sin embargo, no se conocen los mecanismos que expliquen esta relación. Se ha descrito que la insulina regula la transcripción de algunos genes (incluyendo su propia expresión), a través de su interacción con elementos regulatorios presentes en el promotor denominados elementos GAGA28. Una hipótesis interesante es analizar el nivel de expresión de genes regulados por insulina y relacionados ya sea con ND y/o ECV, el que pudiera estar alterado por la presencia de SNPs en dichos elementos. Un análisis bioinformático del promotor del gen de nefrina detectó la presencia de estos elementos GAGA, sin embargo, un estudio piloto en pacientes diabéticos con y sin ND y ECV no detectó alteraciones en estos elementos regulatorios29.
A diferencia de los estudios “gen a gen”, los análisis de GWA han logrado resultados promisorios. En 2007 se publicaron los primeros resultados del proyecto FIND (“Family Investigation of Nephropathy and Diabetes”) que comparan cientos de parejas de hermanos diabéticos con y sin ND de diversos grupos étnicos. Se detectó asociación de las regiones cromosómicas 7q21.3, 10p15.3, 14q23.1 y 18q22.3 con ND y 2q14.1, 7q21.1 y 15q26.3 con albuminuria30. Una segunda publicación de FIND, en la cual se analizan 882 parejas de hermanos diabéticos revela una fuerte asociación entre filtración glomerular y los cromosomas 1q43, 7q36.1, 8q13.3 y 18q23.3. Además en familias mexicanas-americanas se encontró asociación con el cromosoma 2p13.331.
Nuevo nivel de regulación de la expresión génica: microRNA
Una clase importante de elementos reguladores de la expresión génica son los microRNAs (miRNAs), pequeñas cadenas de ácido ribonucleico de 20 a 27 nucleótidos, que modulan múltiples actividades biológicas (Figura 2). Considerados inicialmente como curiosidades en el desarrollo de un nemátodo modelo, en el año 2001 se confirmó su existencia en vertebrados y actualmente han emergido como un nivel de control de genes esencial en todos los genomas de animales y plantas32. Se ha demostrado que estos miRNAs inhiben la expresión de múltiples genes reguladores del desarrollo y proliferación32 y serían importantes en la patogenia y potencialmente una herramienta de diagnóstico de muchas enfermedades. Cada miRNA puede inhibir la expresión de unos pocos a más de 800 genes33, con un promedio de 200 transcritos34, por lo cual estos pequeños RNA resultan ser reguladores maestros de procesos biológicos, modulando la expresión de conjuntos de genes relacionados35. Actualmente, se conocen más de 300 miRNAs humanos distintos, tales como miR-124, miR-let7b, entre otros, los cuales en su conjunto regulan alrededor de 20.000 genes en nuestro genoma. Sin embargo, aún son muy pocos los genes blanco identificados y validados experimentalmente para cada uno de ellos32. A su vez, un mismo gen puede ser regulado por más de un miRNA34.
MicroRNAs y DM2
En 2004 se identificaron miRNAs específicos de páncreas en células de islotes de ratón y de líneas celulares beta y alfa36. El microRNA más abundante encontrado en páncreas se denomina miR-375 y su sobre-expresión inhibe la secreción de insulina mediada por glucosa; la disminución de miR-375 aumenta la secreción de insulina. El mecanismo de acción propuesto es coherente con un efecto a nivel de exocitosis de insulina. Mediante análisis bioinformático se propuso que el mRNA de miotrofina (Mtpn) podría ser un blanco de miR-37536. Los resultados experimentales validan a Mtpn como blanco de este miRNA. La miotrofina sería importante en las etapas finales de la exocitosis de la insulina y el miR-375 (u otros miRNAs) podría ser utilizado para el desarrollo de terapias para DM235. Otro transcrito blanco de miR-375 es el receptor 2 de adiponectina35, lo cual sugiere interesantes conexiones metabólicas, puesto que podría actuar también a nivel de la acción de la insulina (Figura 3).
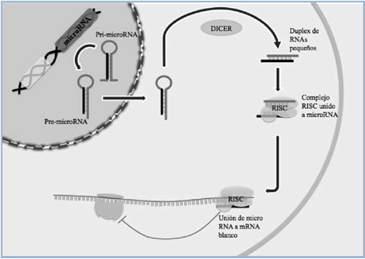 Figura 2. Síntesis de miRNALos miRNAs son codificados por genes que se transcriben generando un RNA precursor primario (pri-miRNA) capaz de plegarse sobre sí mismo. Los pri-miRNAs se procesan a pre-miRNA, los cuales mediante cortes con enzimas específicas (DICER) forman los miRNAs maduros, que ejercen su función regulatoria a nivel post-transcripcional. Los miRNA se unen por complementariedad de bases a mRNAs específicos (secuencias blanco de 6-8 nucleótidos) con la ayuda de complejos proteicos (RISC) y catalizan su degradación o inhiben su traducción. [Adaptado de Gauthier, y Wollheim35].
Figura 2. Síntesis de miRNALos miRNAs son codificados por genes que se transcriben generando un RNA precursor primario (pri-miRNA) capaz de plegarse sobre sí mismo. Los pri-miRNAs se procesan a pre-miRNA, los cuales mediante cortes con enzimas específicas (DICER) forman los miRNAs maduros, que ejercen su función regulatoria a nivel post-transcripcional. Los miRNA se unen por complementariedad de bases a mRNAs específicos (secuencias blanco de 6-8 nucleótidos) con la ayuda de complejos proteicos (RISC) y catalizan su degradación o inhiben su traducción. [Adaptado de Gauthier, y Wollheim35]. 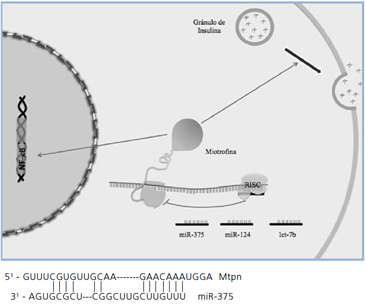 Figura 3. Regulación del mRNA de miotrofina (Mtpn) por miRNA-375A. La exocitosis de insulina requiere de la acción de la miotrofina, cuya traducción es inhibida por miRNA-375. B. La región 3’ no traducida del mRNA de Mtpn correspondiente presenta un sitio único de unión de miRNA-375 por complementariedad de bases perfecta a una secuencia de 7 nucleótidos CUUGUUU y una región de complementariedad imperfecta. [Adaptado de Poy et al.36].
Figura 3. Regulación del mRNA de miotrofina (Mtpn) por miRNA-375A. La exocitosis de insulina requiere de la acción de la miotrofina, cuya traducción es inhibida por miRNA-375. B. La región 3’ no traducida del mRNA de Mtpn correspondiente presenta un sitio único de unión de miRNA-375 por complementariedad de bases perfecta a una secuencia de 7 nucleótidos CUUGUUU y una región de complementariedad imperfecta. [Adaptado de Poy et al.36]. MicroRNAs y ND
En 2007 se demuestra la participacion de un miRNA en la compleja regulacion de un gen relacionado con ND. Este miR-192 se expresa exclusivamente en rinon. Uno de los blancos de miR-192 es el transcrito de SIP1, una proteina que interactua con un factor de transduccion de senales. Al unirse a su blanco, miR-192 inhibe la traduccion de SIP1, que a su vez reprime el gen que codifica la proteina de colageno 1α2. SIP1 ejerce su accion biologica mediante su union a un elemento regulatorio del promotor del gen de colageno 1α2 (E-box)37. De este modo miR-192 regula en forma indirecta la sintesis de colageno 1α2, una proteina de la matriz extracelular que esta aumentada en ND. La expresion de colageno 1α2 no solo es regulada por SIP1, sino que tambien por la proteina represora δEF1, que tambien se une a los mismos elementos de tipo E-box. Una observacion interesante es que miR-192 actua como inhibidor en forma sinergica con δEF1, lo cual indica que los miRNA no solo interactuan con acidos nucleicos, sino que tambien existen interacciones entre microRNA y proteinas37.
MicroRNAs y enfermedad coronaria
La mayoría de las funciones de Angiotensina II (Ang II) son mediadas por AT1R, su receptor, una proteína G que gatilla diversas vías de transducción de señales. Se había descrito que el polimorfismo +1166 A/C en la región 3’ no traducida tenía relación con hipertensión38. Un hallazgo reciente demuestra que la región 3’ es complementaria al miRNA-155, indicando que AT1R es uno de sus genes blanco. La presencia de un polimorfismo en esta secuencia modula la efectividad biológica de la represión ejercida por miR-155, afectando de este modo el número de receptores AT1R presentes en la célula, y por ende la respuesta fisiológica de Ang II39.
Proyecciones futuras
Los estudios de tipo GWA confirman que la base genética de la DM2 y de sus complicaciones corresponde a la participación de múltiples genes. Su mayor aporte consiste en revelar la contribución de diversas regiones cromosómicas y nuevas secuencias relevantes. Una vez identificados los genes localizados en estas regiones, es posible investigar su participación en la etiopatogenia de la DM tipo 2 y de sus complicaciones. El estudio de los microRNA permitió descubrir su capacidad de regular la expresión de sus genes blanco, pero además de interactuar entre ellos o con proteínas regulatorias. Este nuevo escenario revela la infinita complejidad de las redes de regulación de la expresión génica en la célula.
¿De qué manera se podrían aplicar estos nuevos conocimientos a la clínica? En 2005 se publicó un análisis sistemático de perfiles de expresión de todos los miRNAs conocidos en colecciones de tejidos tumorales y líneas celulares representantes de múltiples tipos de tumores40. Para su sorpresa, los autores encontraron que los miRNas presentaban perfiles de expresión diferenciales según el tipo de cáncer. Más aún, se observó un agrupamiento jerárquico según el origen y linaje celular de los tejidos. Este ordenamiento fue mucho más claro que el análisis de perfiles de expresión de genes que codifican para proteínas. Los resultados reportados en este trabajo sugieren que una disminución en la expresión de ciertos miRNAs podría contribuir en forma causal en la generación o mantención de procesos oncogénicos y a su vez surge la posibilidad de utilizar un pequeño número de miRNAs como potentes instrumentos de clasificación y diagnóstico40. Es posible que en un futuro cercano, un análisis similar de la diabetes permita visualizar no sólo un nuevo nivel de comprensión de las bases genéticas de esta compleja enfermedad crónica, sino que el surgimiento de nuevas herramientas de diagnóstico y estrategias terapéuticas.
Referencias
- Mella I, Garcia de los Rios M, Parker M, Covarrubias A. 1981. Prevalencia de la diabetes en el Gran Santiago, Chile. Rev Med Chile 109:869-875.
- Baechler R, Mujica V, Aqueveque X, Ramos L, Soto A. 2002. Prevalencia de diabetes mellitus en la VII Region de Chile. Rev Med Chile 130:1257-1264.
- Lapuz MH. 1997. Diabetic nephropathy. Med Clin of North Am 61:679-88.
- Foley RN, Culleton BF, Parfrey PS, Harnett JD, Kent GM, Murray DC, et al. 1997. Cardiac disease in diabetic end-stage renal disease. Diabetologia 40:1307-1312.
- Gu K, Cowie CC, Harris MI. 1999. Diabetes and decline in heart disease mortality in US adults. JAMA 281:1291-1297.
- Pincus G, White P. 1933. On the inheritance of diabetes an analysis of 675 family histories. Am J Med Sci 186: 1-14.
- Awareness of family health history as a risk factor for disease - United States. 2004. MMWR Morb Mortal Wkly Rep 53:1044-1047.
- Hariri S, Yoon PW, Qureschi N, Valdez R, Scheuner MT, Khoury MJ. 2006. Family history of type 2 diabetes: A population-based screening tool for prevention? Genet Med 8:102-108.
- Pyke DA. 1979. The genetic connections. Diabetologia 17:333-343.
- King H, Aubert RE, Herman WH. 1998. Global burden of diabetes 1995-2025. Diabetes Care 21:1414-1431.
- Santos JL, Perez-Bravo F, Carrasco E, Calvillan M, Larenas G, Albala C. 2001. Low prevalence of type 2 diabetes despite a high average body mass index in the aymara natives from Chile. Nutrition 17:305-309.
- Perez-Bravo F, Carrasco E, Santos JL Calvillan M, Albala C. 2001. Prevalence of type 2 diabetes and obesity in rural mapuche population from Chile. Nutrition 17: 236-238.
- Florez JL, Manning, AK, Dupuis J, McAteer J, Irenze K, Gianniny L et al. 2007. A 100K Genome-Wide-Association Scan for Diabetes and related traits in the Framington Heart Study. Diabetes 56:3063-3074.
- Freedman BI, Tuttle AB, Spray BJ. 1995. Familial predisposition to nephropathy in African-Americans with non-insulin-dependent diabetes mellitus. Am J Kidney Dis 25:710-713.
- Kopyt NP. 2005. Slowing progression along a renal disease continuum. J Am Osteopath Assoc 105:207-215.
- Lewis E, Hunsicker L, Bain R, Rhode R. 1993. The effect of angiotensin- converting-enzyme inhibition on diabetic nephropathy. N Engl J Med 329:1456-1462.
- Susztak K, Bottinger EP. 2006. Diabetic Nephropathy: a frontier for personalized medicine. J Am Soc Nephrol 17:361-367.
- Stoneking, M. 2001. Single nucleotide polymorphisms. From the evolutionary pastc Nature 409:821-822.
- Hirschhorn JN, Daly MJ. 2005. Genome-wide association studies for common diseases and complex traits. Nat Rev Genet 6:95-108.
- Hayes, GM., Pluzhnikov, A, Miyake, K, Sun, Y, Ng, MCY, Roe, CA, et al. 2007. Identification of Type 2 diabetes genes in Mexican americans through Genome-wide association studies. Diabetes 56:3033-3044.
- Wellcome Trust Case Control Consortium. 2007. Genome-wide association study of 14,000 cases of seven common
- Koop K, Eikmans M, Baelde HJ, Kawachi H, de Heer E, Paul LC, et al. 2003. Expression of podocyte-associated molecules in acquired human kidney diseases. J Am Soc Nephrol 14:2063-2071.
- Benigni A, Gagliardini E, Tomasoni S, Abbate M, Ruggenenti P, Kalluri R, et al. 2004. Selective impairment of gene expression and assembly of nephrin in human diabetic nephropathy. Kidney Int 65:2193-2200.
- Lenkkeri U, Mannikko M, McCready P, Lamerdin J, Gribouval O, Niaudet P, et al. 1999. Structure of the gene for congenital nephrotic syndrome of the finnish type (NPHS1) and characterization of mutations. American J Hum Genet 64:51-61.
- Daimon M, Ji G, Oizumi T, Kido T, Baba M, Jimbu Y, et al. 2006. Association of nephrin gene polymorphisms with type 2 diabetes in a Japanese population: The Funagata study. Diabetes Care 29:1117-1119.
- Cartegni L, Wang J, Zhu Z, Zhang MQ, Krainer AR. 2003. ESEfinder: A web resource to identify exonic splicing enhancers. Nucl Acids Res 31: 3568-3571.
- Gonzalez R, Tirado A, Rojas LA, Ossandon F, Alvo M, Wolff C, et al. 2008. Identification and frequency analysis of the putatively pathogenic intronic single nucleotide polymorphism rs#466452 of the nephrin gene in a pilot study of patients with diabetic nephropathy. (manuscrito en revision , enviado a J Diab Compl).
- Kennedy GC, Rutter WJ. 1999. Pur-1, a zinc-finger protein that binds to purine-rich sequences, transactivates an insulin promoter in heterologous cells. Proc Nat Acad Sci 289:11498-11502.
- Gonzalez R, Tirado A, Balanda M, Alvo M, Barquin I, Durruty P, et al. 2007. A pilot study on genetic variation in purine-rich elements in the nephrin gene promoter in type 2 diabetic patients. Biol Res 40:357-364.
- Iyengar SK, Abboud HE, Goddard KA, Saad MF, Adler SG, Arar NH, et al. 2007. Family Investigation of Nephropathy and Diabetes Research Group. Genome-wide scans for diabetic nephropathy and albuminuria in multiethnic populations: the family investigation of nephropathy and diabetes (FIND). Diabetes 56:1577-85.
- Schelling JR, Abboud HE, Nicholas SB, Pahl MV, Sedor JR, Adler SG, et al. 2008. Family Investigation of Nephropathy and Diabetes Research Group. Genome-wide scan for estimated glomerular filtration rate in multi-ethnic diabetic populations: the Family Investigation of Nephropathy and Diabetes (FIND). Diabetes 57:235-43.
- Bartel, DP. 2004. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 116:281-297.
- Rajewsky, N. 2006. MicroRNA target predictions in animals. Nat Genet 38:S8-S13.
- Krek A, Grun D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, et al. 2006. Combinatorial microRNA target predictions. Nat. Genet 37:495-500.
- Gauthier, BR, Wollheim, CB. 2006. MicroRNAs: riboregulators of glucose homeostasis. Nature Med 12:36-29.
- Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, MacDonald PE, et al. 2004. A pancreatic islet-specific microRNA regulates insulin secretion. Nature 432:226-230.
- Kato M, Zhang J, Wang M, Lanting L, Yuan H, Rossi JJ, et al. 2007. MicroRNA-192 in diabetic kidney glomeruli and its function in TGF- ƒÀ-induced collagen expression via inhibition of E-box repressors. Proc Nat Acad Sci 104:3432-37.
- Bonnardeaux A, Davies E, Jeunemaitre X, Fery I, Charru A, Clauser E, et al. 1994. Angiotensin II type 1 receptor gene polymorphisms in human essential hypertension. Hypertension 24:63-69.
- Martin MM, Buckenberger JA, Jiang J, Malana GE, Nuovo GJ, Chotanni M, et al. 2007. The human angiotensin II type 1 receptor +1166 A/C polymorphism attenuates microRNA-155 binding. J Biol Chem 288:24262-24269.
- Lu J, Getz G, Miska EA, Alvarez-Saavedra E, Lamb J, Peck D, et al. 2005. MicroRNA expression profiles classify human cancers. Nature 435:834-838.
Agradecimientos
Agradecemos al Sr. Ares Tirado S. por su colaboración en la confección de las figuras.