Epigenética en obesidad y diabetes tipo 2: papel de la nutrición, limitaciones y futuras aplicaciones
Fermín I. Milagro Y. y J. Alfredo Martínez H.
Epigenetics in obesity and type 2 diabetes: role of nutrition, limitations and future applications
Departamento de Ciencias de la Alimentación y Fisiología, Universidad de Navarra,
Pamplona (España).
CIBERobn, Centro de Investigación Biomédica en Red sobre Fisiopatología de la Obesidad y la Nutrición, Instituto Carlos III,
Madrid (España).
Los autores declaran que no existen situaciones que signifiquen conflicto de intereses.
Correspondencia:
J. Alfredo Martínez
Dpto. de Ciencias de la Alimentación y Fisiología
Universidad de Navarra,
Pamplona, España.
Fax: 0034 948425649
E-mail: jalfmtz@unav.es
Recibido: 28 de mayo de 2013
Aceptado: 19 de junio de 2013
In the last years, epigenetics is helping to explain the mechanisms non dependent on the genetic sequence by which the nutrients and other environmental factors contribute to modulate gene expression and disease development. Obesity and type 2 diabetes are two diseases that have been linked to changes in epigenetic marks (particularly DNA methylation, covalent modifications of histones and miRNAs). These epigenetic changes appear to be influenced, mainly in the perinatal period but also in adulthood, by environmental factors such as hyperglycemia or the diet. Among the food compounds that have been linked to epigenetic modifications, there are methyl donor groups, excessive or deficient caloric intake, short chain fatty acids, some minerals and antioxidant vitamins, and various compounds of plant origin, as catechins, isoflavones or isothiocyanates. EWAS studies, that analyze the methylation of thousands of CpG sites in thousands of individuals, will contribute in the next years to identify some of the epigenetic marks that can be used as early predictors of metabolic risk, as well as some intimate mechanisms that explain the development of obesity, type 2 diabetes and its complications. Moreover, epigenetic marks, among them the CpG-SNPs, can be heritable but some of them could be potentially reversible. One of the medium-term objectives is to develop drug or diet-related treatments that could delay or even reverse these epigenetic changes.
Key words: Epigenetics, histones, DNA methylation, obesity, type 2 diabetes.
Los genes influyen en todos los aspectos de la fisiología humana. La obesidad y la diabetes tipo 2 también son resultado de las interacciones entre la nutrición y el acervo genético. Sin embargo, hasta el momento se ha avanzado poco en el conocimiento de los genes específicos que contribuyen a ambas enfermedades, así como su interacción con los factores ambientales, entre ellos la dieta. En todo caso, se han identificado varias mutaciones que son responsables de formas muy raras, monogénicas, de obesidad (como los portadores de mutaciones en el gen de la leptina)1 y diabetes (las diversas formas de MODY, del inglés “Maturity Onset Diabetes of the Young”)2.
Gracias al desarrollo de los estudios de asociación del genoma completo (en inglés GWAS) se están comenzado a identificar las bases genéticas de las formas comunes de ambas patologías, que están influenciadas por decenas, si no cientos, de genes. En especial, la realización de meta-análisis a gran escala de los resultados de diferentes GWAS se considera en este momento la mejor herramienta a la hora de identificar las variantes genéticas más estrechamente implicadas en el desarrollo de estas enfermedades3,4.
Sin embargo, a pesar de todos estos avances en la genética de las enfermedades metabólicas, estos estudios sólo son capaces de explicar un pequeño porcentaje (alrededor del 10%) de la variabilidad genética respecto a la obesidad. Ante la evidencia del papel que juegan los factores ambientales en el desarrollo de estas patologías, en los últimos años se está investigando la importancia de los procesos epigenéticos. La epigenética se define como aquellas modificaciones heredables de la expresión de los genes que no se encuentran directamente determinadas por la secuencia del ADN5. Se sabe que los factores ambientales pueden provocar modificaciones epigenéticas, que son dependientes de cada tejido y tipo celular, pero se desconoce por el momento la regulación de dichos procesos, la magnitud de los cambios y los tipos celulares en que se producen, los individuos más predispuestos, y las etapas de la vida más cruciales. Otros de los objetivos a largo plazo son identificar marcas epigenéticas que puedan ser utilizadas como predictores tempranos de riesgo metabólico, y desarrollar tratamientos farmacológicos o relacionados con la dieta y el estilo de vida que sean capaces de retrasar dichos cambios epigenéticos e incluso de revertirlos6.
Modificaciones epigenéticas
Muchas modificaciones epigenéticas parecen estar influidas por factores ambientales. En este sentido, la plasticidad del genoma permitiría una mejor adaptación al medio. Los principales mecanismos epigenéticos que regulan la expresión de los genes son:
- La metilación del ADN en citosinas seguidas por guaninas (dinucleótidos CpGs). Estos dinucleótidos son abundantes en las regiones promotoras de muchos genes, y su hipermetilación suele acompañarse de una disminución de la expresión génica 7.
- Diversas modificaciones covalentes en aminoácidos terminales de las histonas, incluyendo, entre otras, acetilación y metilación8, que son moduladas por enzimas como las acetiltransferasas (HATs), metiltransferasas (HMTs), y desacetilasas (HDACs). Estas modificaciones postranscripcionales parecen afectar a la expresión génica a través de alteraciones en el grado de compactación o dificultando el acceso de los factores de transcripción al ADN.
- Los RNAs no codificantes, entre los que destacan los micro RNAs (miRNAs), que regulan post-transcripcionalmente la expresión de genes mediante su emparejamiento con la región no traducida 3 del ARN mensajero y la consiguiente degradación de los transcritos9.
Un hecho de extraordinaria importancia en relación con las marcas epigenéticas es que, al contrario que los polimorfismos genéticos, no son permanentes. Así, diversos factores ambientales, como el estrés, la dieta o el ejercicio físico, y otros relacionados con la obesidad, como la hiperglucemia, el estrés oxidativo, la hipoxia o la inflamación, modulan los cambios epigenéticos y contribuyen a su plasticidad a lo largo de la vida. Finalmente, otros factores originados por la actividad humana, como algunos metales pesados, pesticidas y químicos ambientales, también parecen actuar a través de procesos epigenéticos10. De hecho, este podría ser uno de los mecanismos por los que la exposición a bisfenol A en la época perinatal podría facilitar el desarrollo de diabetes.
Epigenética, época perinatal y heredabilidad
Dos importantes rasgos característicos de los procesos epigenéticos son la capacidad para la transmisión de la memoria celular o heredabilidad transgeneracional, así como la implicación en la diferenciación celular espacial y temporal de las células totipotentes18. La susceptibilidad a cambios en el epigenoma varía en las distintas etapas del ciclo vital, ya que existen ventanas epigenéticas en las que existe mayor predisposición a los cambios. Especialmente sensibles son las épocas fetal y postnatal, aunque también la infancia y la adolescencia son etapas en las que es más probable que se produzcan cambios epigenéticos de cierta magnitud. La epigenética podría ser el eslabón que ayudara a comprender procesos como la programación fetal, la diferenciación de ciertos tipos celulares, las diferencias entre gemelos monocigóticos, y el desarrollo de enfermedades crónicas en el adulto, procesos que interactúan con la ingesta dietética y otros factores ambientales11.
Algunos ejemplos que muestran la importancia de la época perinatal en el desarrollo de obesidad y diabetes en la edad adulta se han centrado en la alimentación materna durante la gestación o el período de lactancia, y en otros factores no nutricionales, como el estrés. Por ejemplo, diversos estudios en humanos y roedores han comprobado que el estrés perinatal (el estrés materno, los bajos niveles de cuidado infantil después del parto y el abuso infantil) afecta a la metilación del ADN del promotor del receptor de glucocorticoides, que se mantiene durante el resto de la vida y se acompaña de una menor expresión de este gen12. En consecuencia, las personas expuestas tienen una mayor respuesta al estrés, lo que lleva a un mayor riesgo de enfermedad, tanto física como psíquica. El receptor de glucocorticoides es clave en la regulación de la adiposidad corporal, especialmente la localizada en la zona visceral, y niveles elevados de glucocorticoides se han ligado al desarrollo de diabetes tipo 2 y obesidad13.
Varios estudios enfocados en la nutrición perinatal han demostrado que la malnutrición calórica o una dieta baja en proteínas durante el embarazo están implicadas en la programación de la descendencia hacia el desarrollo de obesidad y diabetes. Esta desnutrición induce cambios epigenéticos en las vías hipotalámicas fetales que regulan el metabolismo. Por ejemplo, en un estudio con ovejas que sufrieron desnutrición materna moderada14, se redujo la metilación de los promotores de los genes proopiomelanocortina (POMC) y receptor de glucocorticoides (GR) en el hipotálamo fetal, lo que potencialmente puede resultar en una desregulación del balance energético. Estos cambios se asociaron con una menor actividad de la ADN metiltransferasa y una alteración de la metilación y acetilación de las histonas.
En humanos, el estudio del ADN de individuos expuestos prenatalmente a la hambruna holandesa del invierno de 1944-45 mostró cambios numerosos pero relativamente pequeños, que parecían depender en gran medida del momento de la exposición a la hambruna durante la gestación y a su combinación con otras condiciones ambientales. Así, Heijmans et al.15, observaron diferencias sutiles pero persistentes (disminución de ~5%) en la metilación del gen IGF2 de los individuos con exposición prenatal a la hambruna. El análisis de los datos de individuos que sufrieron dicha hambruna, no sólo en la época prenatal sino también postnatal y como adolescentes, muestra que un corto período de desnutrición moderada o severa durante estas edades incrementa el riesgo de diabetes tipo 2 en la edad adulta16.
Aunque este tipo de estudios son muy difíciles de realizar en humanos, numerosos diseños experimentales en animales están investigando la posibilidad de que las marcas epigenéticas inducidas por un período de estrés o malnutrición puedan ser heredadas por las siguientes generaciones, incrementando el riesgo de éstas de sufrir enfermedades metabólicas. Quizás este hecho sea uno de los factores responsables del imparable incremento de la prevalencia de diabetes y obesidad en los últimos decenios. Un estudio muy interesante cruzó entre sí ratas padres y madres que habían sufrido o no diabetes gestacional y analizó la intolerancia a la glucosa durante tres generaciones, así como la expresión de dos genes impresos o imprinted (un tipo particular de regulación epigenética que depende del sexo del progenitor): Igf2 y H1917. El peso al nacer fue significativamente mayor en la descendencia F2 a través de la línea paterna con intolerancia a la glucosa, y a las 3 sem de vida apareció en ellos mayor intolerancia a la glucosa. Igualmente, la expresión de los genes impresos Igf2 y H19 fue menor en los islotes pancreáticos y el esperma de la descendencia F1 y F2, lo que sugiere que los cambios epigenéticos en las células germinales contribuyeron a la transmisión transgeneracional.
Epigenética y obesidad
Estudios recientes muestran que las dietas ricas en grasa y azúcar, así como la obesidad, se asocian con cambios en los patrones de metilación del ADN, que afecta a la región promotora de distintos genes implicados en la homeostasis energética en hígado, como la ácido graso sintasa (FASN) y NDUFB6 18. De manera similar, hay evidencia de que las personas obesas tienen un patrón de marcas epigenéticas diferente del de los individuos no obesos19. Lo difícil, en este caso, es determinar si dichas marcas son producto de la obesidad o alguna de sus complicaciones (resistencia a la insulina, inflamación, estrés oxidativo), de algunos nutrientes que son más o menos frecuentes en la dieta de las personas obesas (algunos ácidos grasos, vitaminas, grupos donantes de metilo como la colina o la metionina), debidos a la exposición a otros factores ambientales o a la herencia. Una de las herramientas que va a ayudar a conocer las características epigenéticas de los sujetos obesos son los estudios de tipo EWAS (estudios de asociación del epigenoma completo, similares a los GWAS), que analizan millones de CpGs en cientos de miles de individuos en el mismo estudio20.
No sólo la ganancia de peso puede estar influenciada por factores epigenéticos. También la pérdida de peso y la restricción energética implican cambios en las marcas epigenéticas, como demostraron Bouchard et al.21, en tres regiones genómicas del tejido adiposo subcutáneo abdominal. Por otra parte, y en relación con el desarrollo de la nutrición personalizada, se han identificado diferentes biomarcadores epigenéticos que parecen predecir el mantenimiento del peso corporal después de la pérdida de peso, como por ejemplo el porcentaje de metilación de las citosinas en el ADN de ATP10A y CD44 en células mononucleares de sangre periférica22. Diferencias similares han sido observadas en otros tejidos, como por ejemplo el tejido adiposo subcutáneo21. En resumen, numerosos estudios sugieren que las diferencias interindividuales en relación con la susceptibilidad a desarrollar obesidad no sólo dependen de la ingesta, el gasto energético y la secuencia genética del individuo, sino también de la herencia epigenética y de influencias nutricionales y de estilo de vida que tiene lugar durante el período intrauterino o ya de adultos, y que modifican las marcas epigenéticas pudiendo afectar a la expresión de los genes.
Algunos genes que sufren regulación epigenética por metilación de las CpGs de su promotor participan en procesos metabólicos clave relacionados con la obesidad, tales como la adipogénesis, el metabolismo energético, el almacenamiento y utilización de lípidos, la inflamación la señalización insulínica y la regulación del apetito. Algunos de ellos se muestran en la Figura 123.
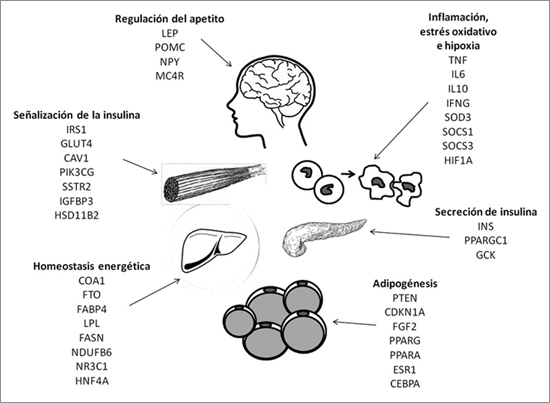
Epigenética y diabetes tipo 2
La diabetes tipo 2 se desarrolla debido a una respuesta inadecuada de las células β pancreáticas y del tejido adiposo frente a un exceso crónico de sustratos energéticos, lo que se traduce en un almacenamiento ectópico de grasa, resistencia a la insulina, concentraciones elevadas de citoquinas inflamatorias y estrés metabólico. Finalmente, conduce a una disminución de la secreción de insulina y a apoptosis de las células β, lo que conlleva una incapacidad para compensar la resistencia a la insulina. Las contribuciones relativas de las células β y del tejido adiposo en el desarrollo de la enfermedad parecen depender de una mezcla de susceptibilidades genéticas y adquiridas (incluyendo cambios epigenéticos en respuesta a estímulos ambientales)24.
Así, un estudio de tipo EWAS que comparó parejas de gemelos monocigóticos de entre 53 y 80 años discordantes para la diabetes tipo 2, identificó 789 sitios CpG diferencialmente metilados en el músculo esquelético y 1.458 en el tejido adiposo subcutáneo25. Aunque la magnitud de esas diferencias no fue muy grande, algunas de ellas se localizaron en genes tan importantes como PPARGC1A y HNF4A. En los islotes pancreáticos, Volkmar et al.26, describieron un número importante de genes aberrantemente metilados en los islotes de pacientes diabéticos que participan en vías relacionadas con la supervivencia y la función de las células β. Estos resultados sugieren que diversos mecanismos epigenéticos pueden estar implicados en la disfunción de las células β y en la patogénesis de la diabetes, abriendo la puerta al estudio del papel de la nutrición, el estrés oxidativo y la inflamación en dichos mecanismos.
Una de las principales prioridades en este campo es la detección temprana de aquellos individuos con más probabilidades de desarrollar diabetes en el futuro. Así, un estudio en células blancas de sangre periférica identificó un sitio CpG en el primer intrón del gen FTO que presentaba una pequeña (3,3%) pero significativa hipometilación en los pacientes diabéticos respecto a los controles27. En una cohorte independiente se reveló que dicha metilación era significativamente menor en los jóvenes que más tarde desarrollaron diabetes tipo 2, con respecto a los jóvenes que no la desarrollaron.
En relación con la resistencia a la insulina, un estudio en 84 parejas de gemelos monocigóticos describió una asociación entre la metilación global del ADN y el índice HOMA28. En este sentido, uno de los genes que puede jugar un papel destacado es PPARGC1A. Así, la metilación del promotor de PPARGC1A aumentó un 50% en los islotes pancreáticos de pacientes diabéticos en comparación con los no diabéticos29. Aunque no se ha descrito una relación clara entre su metilación en el músculo esquelético y la resistencia a la insulina, su metilación en hígado se asocia con resistencia periférica a la insulina y con los niveles de insulina en ayunas30.
Diversos factores ambientales, entre los que destaca la hiperglucemia, contribuyen a la progresión de las complicaciones diabéticas, como por ejemplo la nefropatía, la retinopatía o la microangiopatía. En este sentido, recientes experimentos han revelado una estrecha relación entre los eventos hiperglucémicos (que afectan a la expresión de determinados genes) y ciertas modificaciones en la cromatina. Un ejemplo es la asociación entre los cambios químicos de las colas amino-terminales de la histona H3 y la metiltransferasa Set7 especifica de lisinas. Su expresión parece ser crucial para mejorar la accesibilidad de la cromatina y la transcripción de los genes de las células β, y parece ser responsable de la expresión génica vascular en respuesta a episodios anteriores de hiperglucemia, en lo que se denomina “memoria hiperglucémica”31. De hecho, inhibidores de algunas de las enzimas implicadas en estas reacciones, como las desacetilasas de histonas o HDACs, están siendo investigados como posibles herramientas terapéuticas en la prevención o la reducción de algunas de las complicaciones diabéticas, como la nefropatía32.
Epigenética y nutrición
Entre los factores ambientales que parecen afectar a las marcas epigenéticas se encuentra la dieta. Como ya se ha explicado, una nutrición inadecuada puede originar modificaciones epigenéticas que podrían estar implicadas en un incremento del riesgo a sufrir enfermedades metabólicas. Pero al mismo tiempo, existe la esperanza de que una nutrición adecuada, con un aporte adecuado de compuestos que ayudan a mantener el nivel de metilación del ADN, pueda ayudar a revertir las marcas epigenéticas de riesgo, o prevenir los cambios de metilación que ocurren con la edad o por efecto de otros factores ambientales6.
Los principales factores nutricionales relacionados con la regulación epigenética son los grupos donantes de metilos, en particular los que intervienen en el ciclo de la metionina: metionina, folato, colina, biotina y vitaminas B2, B6 y B1233,34. Estos nutrientes son básicos para mantener los niveles de metilación del ADN y las histonas, siendo la S-adenosín-metionina o SAM la molécula encargada de ceder un grupo metilo a estas macromoléculas. De hecho, una dieta deficiente en grupos donantes de metilo se ha convertido en un modelo habitual de esteatosis hepática que puede acabar desarrollando cirrosis y hepatocarcinoma, mientras que la suplementación con esas mismas moléculas parece revertir la esteatosis mediante cambios en la metilación de genes clave como la ácido graso sintasa35.
También cambios en los porcentajes de macronutrientes y en calorías parecen ejercer un efecto importante en los niveles de metilación del ADN. Estudios en animales han mostrado cambios epigenéticos en modelos de dieta rica en grasa aún sin ir acompañada de un incremento de calorías18. En humanos, la ingesta de una dieta rica en grasa, incluso de forma aguda, indujo cambios en la metilación de 6.508 genes en el músculo esquelético, con un cambio máximo de metilación de 13 puntos porcentuales36. Dichos cambios fueron sólo parcial y no significativamente revertidos tras 6-8 sem de dieta normocalórica, sugiriendo que la reversibilidad de las marcas epigenética es lenta y que la acumulación de modificaciones puede con el tiempo influir en los niveles de expresión de los genes. Este hecho podría estar detrás del conocido como efecto “yo-yo”, que se caracteriza por una dificultad cada vez mayor a adelgazar tras cada episodio de tratamiento hipocalórico.
Pero existen otros factores nutricionales que se han asociado con los cambios epigenéticos, como se muestra en la Figura 2. Entre ellos destaca la suplementación con diversos micronutrientes dietéticos, los ácidos grasos de cadena corta y varios compuestos de origen vegetal con probadas propiedades saludables34.
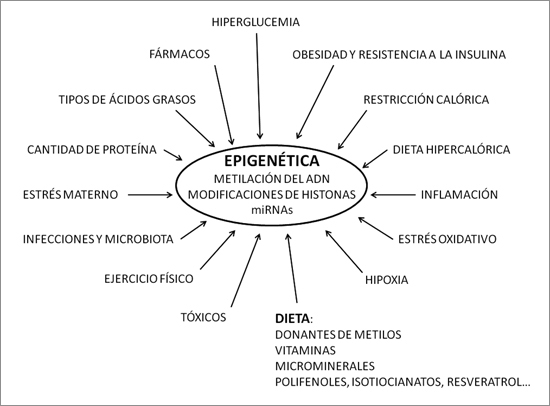 Figura 2. Diversos factores nutricionales que se han asociado con cambios epigenéticos.
Figura 2. Diversos factores nutricionales que se han asociado con cambios epigenéticos.
Conclusiones
La epigenética ayuda a explicar los mecanismos no dependientes de la secuencia genética por los que los nutrientes y otros factores ambientales contribuyen a regular la expresión de los genes. Su naturaleza reversible abre la puerta no sólo a comprender, sino también a tratar enfermedades de origen poligénico y multifactorial, como son la obesidad y la diabetes tipo 2. Un ejemplo de esto es el artículo de Gluckman et al.37, que describe cómo el tratamiento con leptina (entre los días posnatales 3 y 13) de ratas recién nacidas de madres expuestas a un 70% de reducción global de la ingesta calórica durante la gestación, invirtió el fenotipo alterado y la hipermetilación del ADN hepático, aumentando la expresión de PPARα y el receptor de glucocorticoides en ese mismo órgano. Otro campo de interés es el estudio de los CpG-SNPs. Estos son SNPs que introducen o eliminan una citosina susceptible de ser metilada, con lo que no sólo hacen variar la secuencia genética sino también la posible regulación epigenética del gen. Un reciente estudio en islotes pancreáticos de pacientes diabéticos ha destacado que alguno de estos CpG-SNPs estaría relacionado con la expresión del gen e incluso con la secreción de insulina y glucagón38.
Algunos de los principales puntos de interés que se irán dilucidando en los próximos años son:
- La caracterización de los individuos que, a una edad temprana, presentan cambios en los perfiles de metilación de genes específicos (biomarcadores epigenéticos), lo que podría ayudar a predecir su susceptibilidad a desarrollar obesidad o diabetes y prevenir futuras complicaciones.
- El desarrollo y puesta a punto de nuevas herramientas terapéuticas basadas en fármacos o en compuestos presentes en los alimentos que actúen a nivel epigenético.
- Las marcas epigenéticas también podrían tener utilidad como marcadores de pronóstico de la pérdida de peso y en la personalización del tratamiento de la diabetes y de las comorbilidades asociadas a la obesidad.
Por ejemplo, para investigar la importancia del ambiente y la dieta en las modificaciones epigenéticas, una buena estrategia sería comparar animales genéticamente idénticos y que, o bien se han mantenido bajo las mismas condiciones, o bien han sido expuestos a diferentes factores ambientales39.
Sin embargo, la aplicación de la epigenética en la prevención y tratamiento de estas enfermedades todavía está lejos. Falta mucho por conocer acerca de los mecanismos por los que diversos fármacos y compuestos bioactivos presentes en la dieta afectan a los procesos epigenéticos. Una gran dificultad en este ámbito es la falta de especificidad de todos los compuestos conocidos hasta el momento a la hora de influir sobre las marcas epigenéticas en los diversos tipos celulares. Falta también por conocer la importancia real de los procesos epigenéticos sobre el desarrollo de diabetes y obesidad, ya que las magnitudes de los cambios que se observan son sensiblemente inferiores a los que caracterizan a las enfermedades neoplásicas o tumorales.
Aunque la epigenética parece, hoy en día, el candidato perfecto que permite unir los factores ambientales con el desarrollo de problemas metabólicos, incluso años después de haberse producido la exposición a dichos factores, su estudio se enfrenta a una enorme dificultad. Dado que la magnitud del cambio producido por los factores dietéticos y ambientales es pequeña y acumulativa, a que hay muchos factores involucrados, y a que existen múltiples interacciones entre ellos y con la edad, se antoja enormemente difícil desentrañar la importancia individual que tiene cada uno de dichos factores. Esto es más fácil de estudiar in vitro, y ya se están analizando los cambios epigenéticos producidos por diferentes fármacos y compuestos bioactivos en cultivos celulares en situaciones perfectamente controladas. Sin embargo, la mayor dificultad no va a residir en la capacidad para cuantificar las numerosas marcas epigenéticas, ya que las técnicas de microarray y de secuenciación van a permitir medir millones de ellas de manera sencilla. La mayor dificultad la vamos a encontrar a la hora de identificar todos los factores que son capaces de modificar las marcas epigenéticas, así como determinar su importancia real cuando actúan de manera sinérgica o antagónica con cada uno de los demás factores. En este momento no disponemos de herramientas fiables que nos permitan cuantificar, ni siquiera de manera aproximada, todos los factores que afectan a los procesos epigenéticos a los que una persona ha estado expuesta a lo largo de su vida.
Agradecimientos
Los autores agradecen el apoyo económico de la Universidad de Navarra a través de la Línea Especial “Nutrición, Obesidad y Salud”.
Referencias bibliográficas
- Farooqi IS, O’Rahilly S. 2005. Monogenic obesity in humans. Annu Rev Med 56: 443-458.
- Steck AK, Winter WE. Review on monogenic diabetes. 2011. Curr Opin Endocrinol Diabetes Obes 18: 252-258.
- Hale PJ, López-Yunez AM, Chen JY. 2012. Genome-wide meta-analysis of genetic susceptible genes for Type 2 Diabetes. BMC Syst Biol 6: S16.
- Bradfield JP, Taal HR, Timpson NJ, Scherag A, Lecoeur C, Warrington NM, et al; Early Growth Genetics Consortium. 2012. A genome-wide association meta-analysis identifies new childhood obesity loci. Nat Genet 44: 526-531.
- Cordero P, Milagro FI, Campion J, Martínez JA. 2010. Epigenética nutricional: una pieza clave en el rompecabezas de la obesidad. Rev Esp Obes 8: 10-20.
- Milagro FI, Mansego ML, De Miguel C, Martínez JA. 2013. Dietary factors, epigenetic modifications and obesity outcomes: Progresses and perspectives. Mol Aspects Med 34: 782-812.
- García-Carpizo V, Ruiz-Llorente L, Fraga M, Aranda A. 2011. The growing role of gene methylation on endocrine function. J Mol Endocrinol 47: R75-89.
- Fraga MF, Esteller M. 2005. Towards the human cancer epigenome: a first draft of histone modifications. Cell Cycle 4: 1377-1381.
- Zalts H, Shomron N. 2011. The impact of microRNAs on endocrinology. Pediatr Endocrinol Rev 8: 354-362.
- Milagro FI, Mansego ML, De Miguel C, Martínez JA. 2013. Dietary factors, epigenetic modifications and obesity outcomes: Progresses and perspectives. Mol Aspects Med 34: 782-812.
- Martínez JA, Cordero P, Campión J, Milagro FI. 2012. Interplay of early-life nutritional programming on obesity, inflammation and epigenetic outcomes. Proc Nutr Soc 71: 276-283.
- Gudsnuk KM, Champagne FA. 2011. Epigenetic effects of early developmental experiences. Clin Perinatol 38: 703-717.
- Vegiopoulos A, Herzig S. 2007. Glucocorticoids, metabolism and metabolic diseases. Mol Cell Endocrinol 275: 43-61.
- Begum G, Stevens A, Smith EB, Connor K, Challis JR, Bloomfield F, et al. 2012. Epigenetic changes in fetal hypothalamic energy regulating pathways are associated with maternal undernutrition and twinning. FASEB J 26: 1694-1703.
- Heijmans BT, Tobi EW, Stein AD, Putter H, Blauw GJ, Susser ES, et al. 2008. Persistent epigenetic differences associated with prenatal exposure to famine in humans. Proc Natl Acad Sci USA 105: 17046-17049.
- van Abeelen AF, Elias SG, Bossuyt PM, Grobbee DE, van der Schouw YT, et al. 2012. Famine exposure in the young and the risk of type 2 diabetes in adulthood. Diabetes 61: 2255-2260.
- Ding GL, Wang FF, Shu J, Tian S, Jiang Y, Zhang D, et al. 2012. Transgenerational glucose intolerance with Igf2/H19 epigenetic alterations in mouse islet induced by intrauterine hyperglycemia. Diabetes 61: 1133-1142.
- Lomba A, Martínez JA, García-Díaz DF, Paternain L, Marti A, Campión J, et al. 2010. Weight gain induced by an isocaloric pair-fed high fat diet: a nutriepigenetic study on FASN and NDUFB6 gene promoters. Mol Genet Metab 101: 273-278.
- Feinberg AP, Irizarry RA, Fradin D, Aryee MJ, Murakami P, Aspelund T, et al. 2010. Personalized epigenomic signatures that are stable over time and covary with body mass index. Sci Transl Med 2: 49ra67.
- Xu X, Su S, Barnes VA, De Miguel C, Pollock J, Ownby D, et al. 2013. A genome-wide methylation study on obesity: Differential variability and differential methylation. Epigenetics 8 [Epub ahead of print].
- Bouchard L, Rabasa-Lhoret R, Faraj M, Lavoie ME, Mill J, Pérusse L, et al. 2010. Differential epigenomic and transcriptomic responses in subcutaneous adipose tissue between low and high responders to caloric restriction. Am J Clin Nutr 91: 309-320.
- Milagro FI, Campión J, Cordero P, Goyenechea E, Gómez-Uriz AM, Abete I, et al. 2011. A dual epigenomic approach for the search of obesity biomarkers: DNA methylation in relation to diet-induced weight loss. FASEB J 25: 1378-1389.
- Milagro FI, Martínez JA. 2013. Epigenetics of obesity and weight loss. Endocrinol Nutr 60: 12-14.
- Nolan CJ, Damm P, Prentki M. 2011. Type 2 diabetes across generations: from pathophysiology to prevention and management. Lancet 378: 169-181.
- Ribel-Madsen R, Fraga MF, Jacobsen S, Bork-Jensen J, Lara E, Calvanese V, et al. 2012. Genome-wide analysis of DNA methylation differences in muscle and fat from monozygotic twins discordant for type 2 diabetes. PLoS One 7: e51302.
- Volkmar M, Dedeurwaerder S, Cunha DA, Ndlovu MN, Defrance M, Deplus R, et al. 2012. DNA methylation profiling identifies epigenetic dysregulation in pancreatic islets from type 2 diabetic patients. EMBO J 31: 1405-1426.
- Toperoff G, Aran D, Kark JD, Rosenberg M, Dubnikov T, Nissan B, et al. 2012. Genome-wide survey reveals predisposing diabetes type 2-related DNA methylation variations in human peripheral blood. Hum Mol Genet 21: 371-383.
- Zhao J, Goldberg J, Bremner JD, Vaccarino V. 2012. Global DNA methylation is associated with insulin resistance: a monozygotic twin study. Diabetes 61: 542-546.
- Ling C, Del Guerra S, Lupi R, Rönn T, Granhall C, Luthman H, et al. 2008. Epigenetic regulation of PPARGC1A in human type 2 diabetic islets and effect on insulin secretion. Diabetología 51: 615-622.
- Sookoian S, Rosselli MS, Gemma C, Burgueño AL, Fernández Gianotti T, Castaño GO, et al. 2010. Epigenetic regulation of insulin resistance in nonalcoholic fatty liver disease: impact of liver methylation of the peroxisome proliferator-activated receptor γ coactivator 1α promoter. Hepatology 52: 1992-2000.
- Keating ST, El-Osta A. 2012. Chromatin modifications associated with diabetes. J Cardiovasc Transl Res 5: 399-412.
- Advani A, Huang Q, Thai K, Advani SL, White KE, Kelly DJ, et al. 2011. Long-term administration of the histone deacetylase inhibitor vorinostat attenuates renal injury in experimental diabetes through an endothelial nitric oxide synthase-dependent mechanism. Am J Pathol 178: 2205-2214.
- Jiménez-Chillarón JC, Díaz R, Martínez D, Pentinat T, Ramón-Krauel M, Ribó S, et al. 2012. The role of nutrition on epigenetic modifications and their implications on health Biochimie 94: 2242-2263.
- Campión J, Milagro F, Martínez JA. 2010. Epigenetics and obesity. Prog Mol Biol Transl Sci 94: 291-347.
- Cordero P, Gómez-Uriz AM, Campion J, Milagro FI, Martínez JA. 2013. Dietary supplementation with methyl donors reduces fatty liver and modifies the fatty acid synthase DNA methylation profile in rats fed an obesogenic diet. Genes Nutr 8: 105-113.
- Jacobsen SC, Brøns C, Bork-Jensen J, Ribel-Madsen R, Yang B, Lara E, et al. 2012. Effects of short-term high-fat overfeeding on genome-wide DNA methylation in the skeletal muscle of healthy young men. Diabetología 55: 3341-3349.
- Gluckman PD, Lillycrop KA, Vickers MH, Pleasants AB, Phillips ES, Beedle AS, et al. 2007. Metabolic plasticity during mammalian development is directionally dependent on early nutritional status. Proc Natl Acad Sci USA 104: 12796-12800.
- Dayeh TA, Olsson AH, Volkov P, Almgren P, Rönn T, Ling C. 2013. Identification of CpG-SNPs associated with type 2 diabetes and differential DNA methylation in human pancreatic islets. Diabetología 56: 1036-1046.
- Moleres A, Marti A. 2008. Influencia del ambiente y la alimentación en la programación epigenética de la obesidad. Rev Esp Obesidad 6: 66-74.