microRNAs y diabetes tipo 1
Diego F. García-Díaz1,a y Francisco Pérez B.1,b
microRNAs and type 1 diabetes
1Laboratorio de Nutrigenómica.
Departamento de Nutrición.
Facultad de Medicina. Universidad
de Chile.
aBiotecnólogo, PhD en Nutrición.
bBioquímico, PhD en Ciencias
Biológicas.
Correspondencia a:
Dr. Francisco Pérez Bravo
Av. Independencia 1027, Santiago.
Teléfono: + 56 2 2978 61 35
fperez@med.uchile.cl
Recibido: 03-08-2016
Aceptado: 22-09-2016
The worldwide increased incidence of type 1 diabetes (T1D) and the decreased genotypes that confer increased risk to T1D indicate a strong environmental impact on the disease. These mechanisms could occur through epigenetic modifications that operate on several gene expression patterns (methylation and acetylation, among others). An alternative mechanism of gene expression inhibition are the microRNAs families. These small noncoding RNAs bind the of mRNAs, downregulating and can downregulate the expression of multiple genes. In this review we discussed the role of certain miRNAs in three characteristics observed in T1D, such as inflammation, autoimmunity and apoptosis.
Key words: Type 1 diabetes, epigenetics, miRNAs.
La diabetes mellitus tipo 1 (DM1) es el resultado
de la destrucción autoinmune de las células β
pancreáticas en más del 90% de los casos nuevos.
Antes de la aparición de los síntomas de la enfermedad,
las personas susceptibles pueden o no presentar
anticuerpos contra proteínas del islote pancreático (estado
de pre-diabetes). A pesar de disponer de una diversidad
de auto-anticuerpos como marcadores de la patología
(GAD65, IA-2, IAA, ICA, y ZnT8) estos tienen un bajo
poder predictivo. La mayoría de los casos se diagnostican
en la infancia y adolescencia, y la terapia con insulina
es hasta el momento el único tratamiento disponible1.
Entre los factores ambientales que están relacionados
como posibles iniciadores de la DM1 o aceleradores del
proceso autoinmune se describen: a la dieta o la introducción
temprana de leche de vaca, algunos agentes virales
(enterovirus, rotavirus) y el exceso de higiene en
edades tempranas (hipótesis de la higiene)2-4.Desde el
punto de vista genético, los estudios familiares muestran
una agregación del componente genético en familiares de
primer grado. Los hermanos de un pacient con DM1 tienen
hasta 15 veces mayor riesgo en comparación con la
población general, y este riesgo se incrementa a 50 para
gemelos monocigóticos. Esta agregación apoya la existencia
de la susceptibilidad genética en la DM1 y en la
actualidad, se han identificado más de 20 loci que podrían
estar participando5. El más importante de ellos continúa
siendo la región del complejo mayor de histocompatibilidad
(MHC; cromosoma 6; locus llamado IDDM1) que
aporta aproximadamente el 50% de la heredabilidad en
la DM1. La asociación más fuerte con la enfermedad se
encuentra en los genes MHC de clase II (DR y DQ) que
participan en la presentación antigénica. En concreto, los
haplotipos DRB1*03-DQA1*0501-DQB1*0201 (DR3)
y DRB1*04-DQA1*0301-DQB1*0302 (DR4) confieren
el mayor riesgo, mientras que DRB1*15-DQA1*0102-
DQB1*0602 (DR2) otorgarían protección contra la enfermedad6-
7. Alrededor de un 70% de los DM1 poseen la
combinación DR3/DR4, sin embargo, esta combinación
se encuentra en un 30-40% de la población sana, lo que
demuestra que esa región genética otorga una importante
susceptibilidad, pero que no es absoluta. La hipótesis más
aceptada indica que, además de la región HLA, existe la
contribución de un gran número de genes no-HLA que incrementan
el riesgo para desarrollar DM1. Entre ellos se
describe a los genes CTLA-4, PTPN22, PDCD1, VNTRINS,
entre otros8. Varios de estos genes están asociados
a la inmuno-regulación negativa, es decir, colaboran frenando
una respuesta inmune ya activada. Junto a la presentación
de antígenos por medio del complejo HLA de
clase II, parecen cumplir un papel esencial en los procesos
de infiltración de células T en el islote β pancreático.
microRNAs y DM1
Como ya se ha descrito, la base genética de la DM1 no tiene un patrón de herencia mendeliana, por lo tanto, el mejor modelo para explicar la susceptibilidad implica un número diverso de genes que confieren pequeños efectos sobre el riesgo9. Además, debido a la heterogeneidad de estos genes, algunos loci pueden tener fuertes efectos sobre una población particular o un subconjunto de familias y no tener ningún efecto en otro grupo. De este modo se entiende que la DM1 se aproxima más a un modelo epigenético, donde existe un mayor nivel de complejidad a la observada considerando sólo las variaciones genéticas (polimorfismos). Dentro de los mecanismos epigenéticos se encuentran los miRNAs10. Los miRNAs son una clase de ARNs no codificantes de una sola hebra (21-25 nt) que se transcriben a partir de ADN, pero que no se traducen en proteínas, y que se cree que funcionan, al menos en animales, mediante la inhibición de la traducción del ARNm a través de apareamientos imperfectos en la región 3 ‘no traducida (3’ UTR) de estos11. El primer indicio de que los miRNAs podrían estar involucrados en la regulación de la función de las células β fue la identificación de dos miRNAs expresados específicamente en islotes pancreáticos humanos: miR-375 y miR-37612. El miR-375 regula negativamente la secreción de insulina estimulada por glucosa, controlando principalmente la expresión de miotrofina y también se encuentra relacionado con la regulación de la vía de señalización de PI3K. Desde entonces, se han descrito una serie de miRNAs que son capaces de regular la función β pancreática (miR-9, miR-410, miR-130a)13,14, desarrollo del páncreas15 y regeneración de la célula β pancreática16. Si bien hay escasa evidencia al respecto, estos estudios presentan buena evidencia de cómo los miRNAs pueden presentar un papel importante en la serie de eventos moleculares que ocurren en el páncreas. Estudios bioinformáticos entre las regiones cromosómicas de 530 miRNAs con genes de susceptibilidad a DM1 mostró la existencia de 27 miRNAs que se encuentran en loci humanos asociados con DM1. Curiosamente, los blancos objetivos previstos para estos miRNAs incluyen genes relacionados con la autoinmunidad, con células β, moléculas co-estimuladoras de células T y CD28 (miR-16-2), INFγ y FasL (miR-551b, miR-877) y secreción de insulina (miR-375). Como cada miRNA puede dirigirse a múltiples mRNAs, a menudo en combinación con otros miRNAs, estas moléculas crean complejas redes reguladoras de la expresión génica17. La exposición crónica a citoquinas pro-inflamatorias induce cambios en la expresión de los genes causando alteración en la vía de señalización de dichos genes18. Reportes recientes han demostrado que la exposición de células β a citoquinas pro-inflamatorias produce cambios en la expresión de tres microRNAs: miR-21, miR-34 a y miR- 146; produciendo alteraciones en la producción de insulina y en la apoptosis, encontrándose que los niveles de expresión de estos miRNAs están aumentados en islotes de ratones diabéticos19.
microRNAs e inflamación
El papel de los miRNA en diversas enfermedades humanas (por ejemplo, cáncer, enfermedades autoinmunes e inflamación) puede relacionarse a través de diferentes mecanismos, como la inactivación epigenética. La expresión de un miRNA puede ser inducida por una variedad de estímulos como el estrés celular, la hiperglicemia o la inflamación. Tanto la inducción como la represión de los miRNAs en respuesta a diferentes estímulos puede influir en procesos biológicos con efectos pro o antiinflamatorios. Se han descrito diversos miRNAs que podrían actuar como mediadores de la inflamación a través de la regulación de genes de quimioquinas. Entre los que se han descrito, destacan miR-132, miR-146 y miR-155, que pueden activarse por mediadores inflamatorios, incluyendo los componentes de la ruta NF-κβ. Estudios recientes han demostrado que en macrófagos se expresa miR-155 en respuesta a TNF-α e IFN-β. A su vez, miR-146a y miR-146b son capaces de inhibir al receptor de IL-1 y TNF-α. También se ha descrito que miR-9 se sobre-expresa en neutrófilos y monocitos después de la activación de TLR4, luego del estímulo con TNF-α, IL-1β, pero no por IFN-γ. Otros múltiples miRNAs han sido asociados a respuesta inflamatoria como miR-21, miR-147, miR-513 y miR-98, evidenciando el papel de los miRNAs como importantes reguladores de la inflamación20- 21.
La exposición prolongada a IL-1β o TNF-α produce aumento en la expresión de miR-21, miR-34, y miR-146 a en células β pancreáticas de ratones19, lo que se corrobora con otros estudios realizados en miR-29 que demostraron que la exposición prolongada a citoquinas aumentó la expresión de miR-29 en células β, la misma que se acompañó de la baja regulación de las proteínas anti-apoptóticas lo que desencadenó la apoptosis de dichas células22. Se ha demostrado que la estimulación mediante TNF-α induce la expresión de algunos miRNAs, incluyendo a miR-146a y miR-155, lo que afecta la patogénesis de algunas enfermedades como la Artritis Reumatoide23.
Esto sugiere que miR-155 tiene un papel como regulador
de la inflamación; sin embargo, cuando se analizó
a otro microRNA, como miR-146a, se observó que se activó con concentraciones mucho más bajas de LPS (0,1
ng/ml) lo que sugiere que este miRNAs es mucho más
sensible a cambios en su entorno24. miR-146ª juega un rol importante como un regulador negativo de inflamación,
proliferación de células mieloides y del cáncer25. La baja
expresión de miR-146a en ratones produce el aumento
excesivo de citoquinas pro inflamatorias (TNF-α and IL-
6) en respuesta a LPS25. El “knockout” de miR-146a en
ratones produce esplenomegalia, linfoadenopatías, inflamación
multi-orgánica y daño tisular, que se acompaña de
niveles altos de anticuerpos y de citoquinas pro inflamatorias,
lo que sugiere la participación de este microRNA en
el control del sistema inmune26.
microRNAs y autoinmunidad
Se conoce que algunos miRNAs están relacionados en el desarrollo y función de las células inmunes maduras. miR-155 ha mostrado ser un factor clave para la respuesta inmune innata y adquirida ya que es necesario para la diferenciación y función de las células T. Ensayos en ratones deficientes en miR-155 muestran inmunodeficiencia y defectos en la función las células dendríticas, células B y linfocitos T. miR-150, que es expresado específicamente en los linfocitos maduros, puede bloquear el desarrollo de células β cuando se expresa tempranamente. miR-181 está implicado en la sensibilidad de las células T en respuesta a antígenos a través de regulación de fosfatasas en la vía de señalización del receptor de células T y miR-223 está relacionado a la regulación en la maduración de granulocitos y la respuesta inflamatoria27.
Estudios de perfiles de expresión han demostrado una desregulación de la expresión de los genes de miRNAs en diversos tumores y también se ha visto que algunos miRNAs como el miR-15 o miR-16 podrían tener funciones como oncogenes o como supresores de oncogenes, regulando genes pro-apoptóticos como BCL-2 que está altamente expresado en leucemia linfocítica crónica28. Algunos miRNAs han sido involucrados con la autoinmunidad y la destrucción de las células β, mediante el cambio de expresión de éstos en células T de individuos afectados con DM1. Se ha observado aumento en la expresión de miR-510 y baja expresión en miR-191 y miR-342 en pacientes con DM1 con respecto a individuos sanos, lo que sugiere el rol de los miRNAs en enfermedades autoinmunes30.
Por otra parte, se han identificado miRNAs que están
implicados en la regulación del sistema inmune cuyas
alteraciones están directamente relacionadas con desórdenes
inmunológicos. Por ejemplo, miR-146a, miR-155
y miR-16, se han encontrado alterados en linfocitos T
de pacientes con Artritis Reumatoide (AR)29 y se ha encontrado
que miR-326 se encuentra alterado en células
mononucleares periféricas de pacientes con Esclerosis
Múltiple31.
microRNAs y apoptosis
El papel de los miRNAs en la señalización apoptótica aún no se ha determinado totalmente, sin embargo, varios estudios muestran que podrían tener un papel preponderante en este proceso.
La participación de los miRNAs en la muerte celular se informó por primera vez cuando se mostró que miR-14 regulaba la apoptosis en la Drosophila. miR-15a y miR- 16 inducen apoptosis mediante la participación de Bcl2. También miR-15a y miR-16 se encuentran significativamente reducidos en el 68% de los pacientes con Leucemia Linfocítica Crónica32. Trabajos realizados por Cimmino y cols sugieren que estos dos miRNAs antagonizan el desarrollo del tumor mediante regulación de la apoptosis. Los autores demostraron que la expresión de miR-15a y miR-16 tienen un efecto inverso sobre la expresión de Bcl2 en células provenientes de pacientes con Leucemia Linfocítica Crónica33.
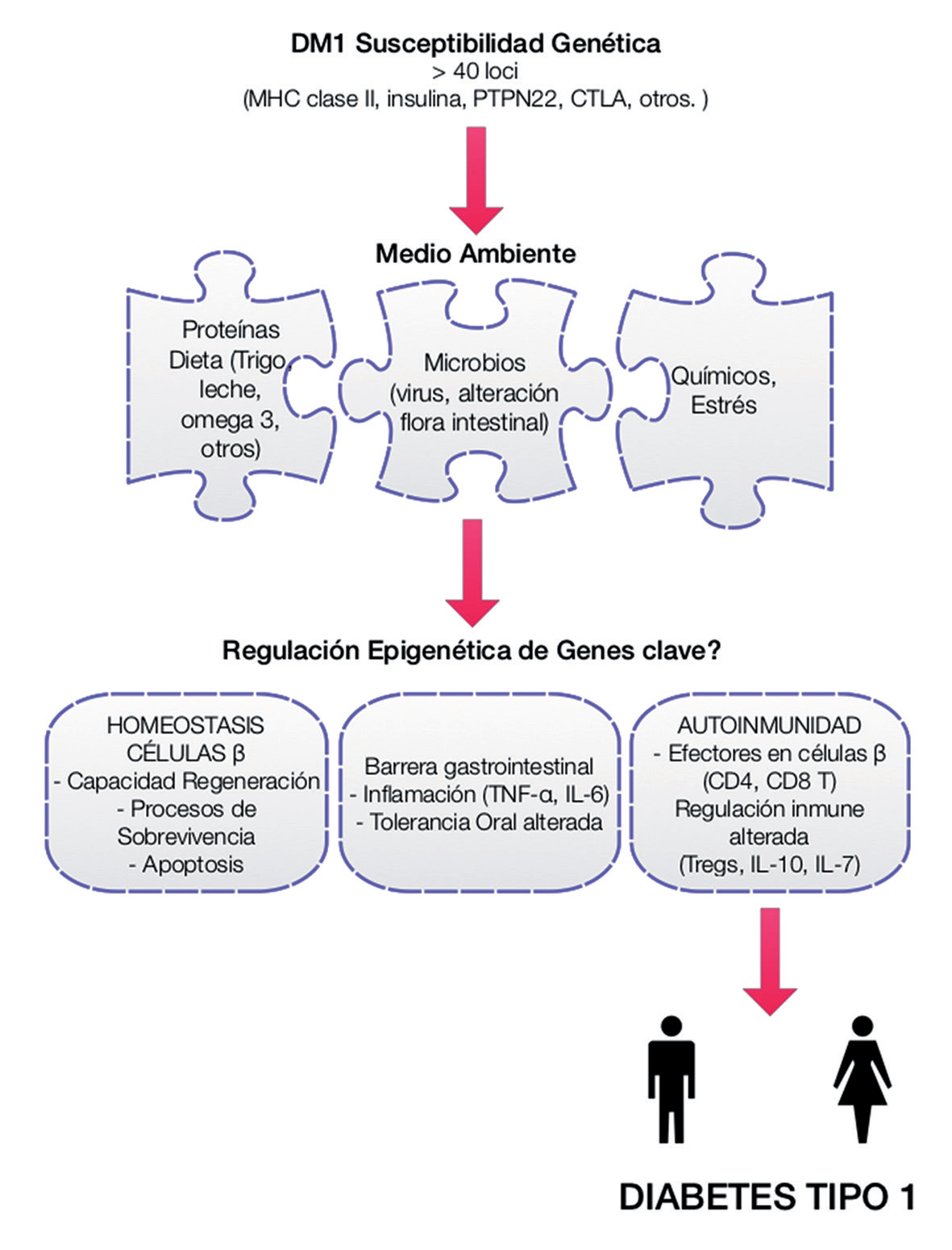
1.- Factores genéticos y ambientales en la etiología de la DM1, escenarios de regulación epigenética. Adaptado de MacFarlane, 2009.
miR-15 y miR-16 se han asociado a la regulación de
Bcl-2. Esta molécula pertenece a la familia de proteínas
que regulan la apoptosis celular y que contiene los
miembros pro-apoptóticos y anti-apoptóticos, todos los
cuales actúan como reguladores fundamentales en la vía
intrínseca. La vía intrínseca de apoptosis se desencadena
a través de diversos estímulos tales como fármacos citotóxicos
y el estrés oxidativo. Se ha demostrado una correlación
inversa entre miR-15 y miR-16 con los niveles
de proteína Bcl-2. La transfección de miR-15 y miR-16,
da como resultado una reducción completa en la expresión
de Bcl-2. Esto ha sido corroborado desde el punto
de vista de la fragmentación del ADN. En conjunto, estos
datos indican claramente el miR-15 y miR-16 son
reguladores de Bcl-2 y podrían explicar el mecanismo
de la sobreexpresión de Bcl-2 en un número importante
de cánceres humanos. La inducción de apoptosis en la
vía Bcl-2 por miR-15 y miR-16 in vitro indica que la
desregulación de estos miRNAs es un evento clave en la
evasión de la apoptosis33. Sin embargo, nuestros resultados
recientes no han encontrado para DM1 una relación
significativa entre estos miRNAs y otros que se han investigado,
y el fenómeno de apoptosis34,35.
En resumen, existe importante evidencia que apoya
el papel de los miRNAs en distintos ámbitos tales como
la iniciación y progresión de ciertos tipos de cáncer, la
respuesta inmune, la autoinmunidad, la proliferación celular,
la muerte celular y la inflamación, todos fenómenos
regulados directa o indirectamente vía el factor NF-κβ.
De tal modo que resulta plausible establecer posibles
convergencias entre determinados miRNAs y sus genes
blancos con las vías de señalización asociadas a NF-κβ
en enfermedades que comparten mecanismos asociados
a la autoinmunidad, apoptosis e inflamación, todos fenómenos
que se observan en las distintas fases de la DM1,
tanto al inicio, como durante su progresión.
Agradecimientos
Esta publicación es parte del proyecto FONDECYT
1130240 (Dr. Francisco Pérez Bravo).
Referencias bibliográficas
- Atkinson MA. 2012. The pathogenesis and natural history of type
1 diabetes. Cold Spring Harb Perspect Med 2 (1 1): 1-18.
- Rønningen KS. 2015. Environmental trigger (s) of type 1 diabete s:
why so difficult to identify? Biomed Res Int 321656.
- Kagohashi Y, Otani H. 2015. Role of nutritional factors at the
early life stages in the pathogenesis and clinical course of type 1
diabetes. Biomed Res Int 382165.
- Lönnrot M, Lynch K, Larsson HE, Lernmark A, Rewers M,
Hagopian W, She JX, et al. 2015. A method for reporting and
classifying acute infectious diseases in a prospective study of
young children: TEDDY. BMC Pediatr 20: 15-24.
- Nokoff N, Rewers M. 2013. Pathogenesis of type 1 diabetes:
lessons from natural history studies of high-risk individuals. Ann
N Y Acad Sci 1281: 1-15.
- Pugliese A. 2013. The multiple origins of Type 1 diabetes. Diabet
Med 30 (2): 46-135.
- Egro FM. 2013. Why is type 1 diabetes increasing? J Mol
Endocrinol 51 (1): 1-13.
- Noble JA. Immunogenetics of type 1 diabetes: A comprehensive
review. J Autoimmun 2015; 64: 101-112.
- Hewagama A, Richardson B. 2009. The genetics and epigenetics of
autoimmune diseases. J Autoimmun 33 (1): 3-11.
- Stankov K, Benc D, Draskovic D. 2013. Genetic and epigenetic
factors in etiology of diabetes mellitus type 1. Pediatrics 132 :
1112-1122.
- Flynt AS, Lai EC. 2008. Biological principles of microRNAmediated
regulation: shared themes amid diversity. Nature Rev
Genet 9 (11): 831-842.
- Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X,
Macdonald PE, Pfeffer F, et al. 2004. A pancreatic
islet-specific microRNA regulates insulin secretion.
Nature 432: 226-230.
- Hennessy E, Clynes M, Jeppesen PB, O'Driscoll L. 2010.
Identification of microRNAs with a role in glucose stimulated
insulin secretion by expression profiling of MIN6 cells. Bioche m
Biophys Res Commun 396: 457-462.
- Plaisance V, Abderrahmani A, Perret-Menoud V, Jacquemin
V, Lemaigre F, Regazzi R. 2006. MicroRNA-9 controls the
expression of Granuphilin/Slp4 and the secretory response of
insulin-producing cells. JBC 281: 26932-26942.
- Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD,
Plasterk RH. 2007. Targeted inhibition of miRNA maturation
with morpholinos reveals a role for miR-375 in pancreatic islet
development. PLoS biology 5: e203.
- Bonner-Weir S, Li WC, Ouziel-Yahalom L, Guo L, Weir GC,
Sharma A. 2010. Beta-cell growth and regeneration: replication is
only part of the story. Diabetes 59: 2340-2348.
- Klein D, Misawa R, Bravo-Egana V, Vargas N, Rosero S, Piroso J,
Ichii H, et al. 2013. MicroRNA expression in alpha and beta cells
of human pancreatic islets. PLoS One 8 (1): e55064.
- Eizirik D, Colli M, Ortis F. 2009. The role of inflammation in
insulitis and beta cell loss in type 1 diabetes. Nat Rev Endocr inol
5: 219-226.
- Roggli E, Britan A, Gattesco S, Lin-Marq N, Abderrahmani A, Meda P, Regazzi R. 2010. Involvement of microRNAs in the cytotoxic effects exerted by proinflammatory cytokines on pancreatic beta-cells. Diabetes 59 (4): 978-986.
- Guay C, Regazzi R. 2013. Circulating microRNAs as novel
biomarkers for diabetes mellitus. Nat Rev Endocrinol 9 (9):
513-521.
- Taganov K, Boldin M, Chang K. 2006. NFk-B-dependent
induction of microRNA miR-146, an inhibitor targeted to signaling
proteins of innate immune responses. Proc Natl Acad Sci USA
103: 12481-12486.
- Allagnat F, Cunha D, Moore F, Vanderwinden JM, Eizirik DL,
Cardozo AK. 2011. Mcl-1 downregulation by pro-inflammatory
cytokines and palmitate is an early event contributing to β-cel l
apoptosis. Cell Death Differ 18 (2): 328-337.
- Pauley KM, Satoh M, Chan AL, Bubb MR, Reeves WH, Chan
EK. 2008. Upregulated miR-146a expression in peripheral blood
mononuclear cells from rheumatoid arthritis patients. Arthritis Res
Ther 10 (4): R101.
- Schulte LN, Westermann AJ, Vogel J. 2013. Differential
activation and functional specialization of miR-146 and
miR-155 in innate immune sensing. Nucleic Acids Res 7 41 (1):
542-553.
- Boldin MP, Baltimore D. 2012. MicroRNAs, new ef fectors and
regulators of NF-κB. Immunol Rev 246 (1): 205-220.
- Zhao JL, Rao DS, Boldin MP, Taganov KD, O'Connell RM,
Baltimore D. 2011. NF-kappaB dysregulation in microRNA-146adeficient
mice drives the development of myeloid malignancies.
Proc Natl Acad Sci USA 08 (22): 9184-9189.
- Simpson LJ, Ansel KM. 2015. MicroRNA regulation of
lymphocyte tolerance and autoimmunity. J Clin Invest 125 (6):
2242-2249.
- Calin GA, Cimmino A, Fabbri M, Ferracin M, Wojcik SE,
Shimizu M, Taccioli C, et al. 2008. MiR-15a and miR-16-1
cluster functions in human leukemia. Proc Natl Acad Sci
USA 105: 5166-5171.
- Stanczyk J, Pedrioli DM, Brentano F, Sánchez-Pernaute O,
Kolling C, Gay RE, Detmar M, et al. 2008. Altered expression
of MicroRNA in synovial fibroblasts and synovial tissue in
rheumatoid arthritis. Arthritis and rheumatism 58: 1001-1009.
- Hezova R, Slaby O, Faltejskova P, Mikulkova Z, Buresova I,
Raja KR, Hodek J, et al. 2010. microRNA 342, microRNA-191
and microRNA-510 are differentially expressed in T regulatory
cells of type 1 diabetic patients. Cell Immunol 260 (2): 70-74.
- Du C, Liu C, Kang J, Zhao G, Ye Z, Huang S, Li Z, et al. 2009.
MicroRNA miR-326 regulates TH-17 differentiation and is
associated with the pathogenesis of multiple sclerosis. Nature
immunology 10: 1252-1259.
- Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Noch E,
et al. 2002. Frequent deletions and down-regulation of micro-
RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic
leukemia. Proc Natl Acad Sci USA 99: 15524-15529.
- Cimmino A, Calin GA, Fabbri M, Iorio MV, Ferracin M,
Shimizu M, et al. 2005. miR-15 and miR-16 induce apoptosis by
targeting BCL2. Proc Natl Acad Sci USA 102: 13944-13949.
- Salas-Pérez F, Codner E, Valencia E, Pizarro C, Carrasco E,
Pérez-Bravo F. 2013. MicroRNAs miR-21a and miR-93 are down
regulated in peripheral blood mononuclear cells (PBMCs) from
patients with type 1 diabetes. Immunobiology 218: 733-737.
- Estrella S, García-Díaz D, Codner E, Camacho-Guillén P, Pérez-Bravo F. 2016. Expresión de miR-22 y miR-150 en diabetes mellitus tipo 1: posible asociación con autoinmunidad y características clínicas de la enfermedad. Med Clin (Barc) 147 (6): 245-247.